El diagnóstico de enfermedades infecciosas tiene como principal objetivo identificar el agente o los agentes causantes para poder aplicar el tratamiento más eficaz, así como establecer las medidas de prevención adecuadas.
Para tal fin, existen diversas técnicas o metodologías que permiten determinar la presencia del agente patógeno de forma directa (por cultivo microbiológico o por técnicas moleculares) o indirecta (detección de anticuerpos mediante técnicas serológicas). Además, en muchas ocasiones no siempre es suficiente con identificar la especie que está causando el problema sino llegar a su caracterización a nivel de subespecie.
TOMA DE MUESTRAS
La obtención de resultados de calidad, es decir, fiables y representativos del proceso de la explotación, está supeditada a una correcta selección de animales y muestras a tomar.
A la hora de seleccionar los animales se debe tener en cuenta:
 Analizar más de un animal para que los resultados sean representativos del grupo de animales afectados.
Analizar más de un animal para que los resultados sean representativos del grupo de animales afectados.
 Enviar muestras antes de instaurar el tratamiento antibiótico, ya que éste interfiere en los resultados microbiológicos.
Enviar muestras antes de instaurar el tratamiento antibiótico, ya que éste interfiere en los resultados microbiológicos.
 Enviar muestras de animales sacrificados o en su defecto recién muertos, ya que la autolisis de las muestras afecta en gran medida al éxito diagnóstico.
Enviar muestras de animales sacrificados o en su defecto recién muertos, ya que la autolisis de las muestras afecta en gran medida al éxito diagnóstico.
 Al seleccionar el número de muestras, es importante estratificarlas en función de la edad de los animales, del estado productivo etc.
Al seleccionar el número de muestras, es importante estratificarlas en función de la edad de los animales, del estado productivo etc.
El tipo de muestra se seleccionará en función de[registrados]:
 El tipo de proceso: reproductivo, digestivo, respiratorio, cutáneo, articular, poliserositis o nervioso.
El tipo de proceso: reproductivo, digestivo, respiratorio, cutáneo, articular, poliserositis o nervioso.
 El agente patógeno a estudiar.
El agente patógeno a estudiar.
 La técnica diagnóstica solicitada: cultivo microbiológico, qPCR, serología, etc.
La técnica diagnóstica solicitada: cultivo microbiológico, qPCR, serología, etc.
 El objetivo del análisis: monitorización o diagnóstico
El objetivo del análisis: monitorización o diagnóstico
Se recomienda consultar previamente con el laboratorio. En la Tabla 1 se recogen los principales tipos de muestras en función del proceso a estudiar.
CULTIVO MICROBIOLÓGICO Y PRUEBAS DE SENSIBILIDAD ANTIBIÓTICA
IDENTIFICACIÓN Y CARACTERIZACIÓN DE PATÓGENOS
El cultivo microbiológico es la técnica clásica para diagnóstico de enfermedades infecciosas bacterianas. Consiste en cultivar la muestra clínica en medios de crecimiento específicos y la posterior identificación de las colonias con interés clínico obtenidas.
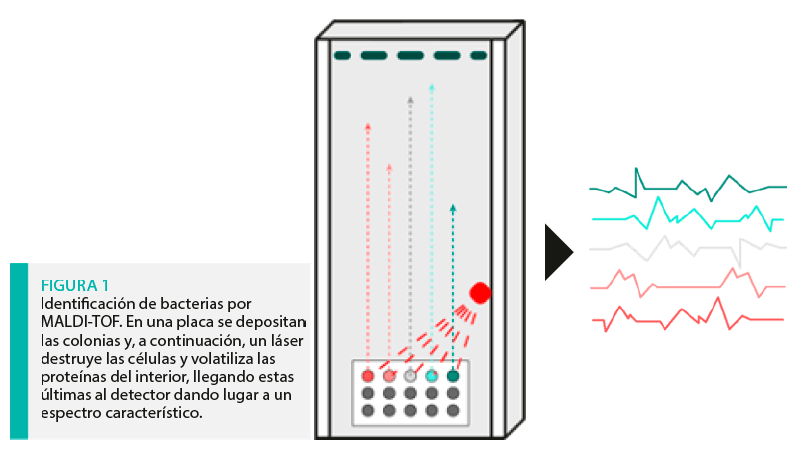
La identificación y caracterización de las colonias se ha realizado tradicionalmente por pruebas bioquímicas, aunque en los últimos años, el uso de tecnologías como la espectrometría de masas -equipos MALDI-TOF (Matrix-Assisted Laser Desorption/Ionization – desorción/ionización láser asistida por matriz)- han cobrado una gran relevancia para este propósito. Este tipo de análisis destaca por la rapidez y la identificación fiable que proporciona a nivel de género y especie.
La tecnología MALDI-TOF permite detectar el conjunto de proteínas de las distintas especies bacterianas ya que cada una de ellas posee una “huella molecular” característica. Estas huellas se comparan con una base de datos con miles de entradas de bacterias patógenas en veterinaria para saber de qué bacteria se trata (Figura 1).
ANTIBIOGRAMA Y CONCENTRACIÓN MÍNIMA INHIBITORIA
Los aislamientos bacterianos de interés que pueden estar causando el proceso clínico deben someterse a pruebas de sensibilidad antibiótica, siendo de especial importancia dada la creciente problemática de la aparición de resistencias.
Las pruebas de sensibilidad antibiótica, entre las que destacan el método de Kirby Bauer (disco-placa) o concentración mínima inhibitoria (CMI), permiten determinar el perfil de sensibilidad de las cepas analizadas y decidir la mejor opción de tratamiento.
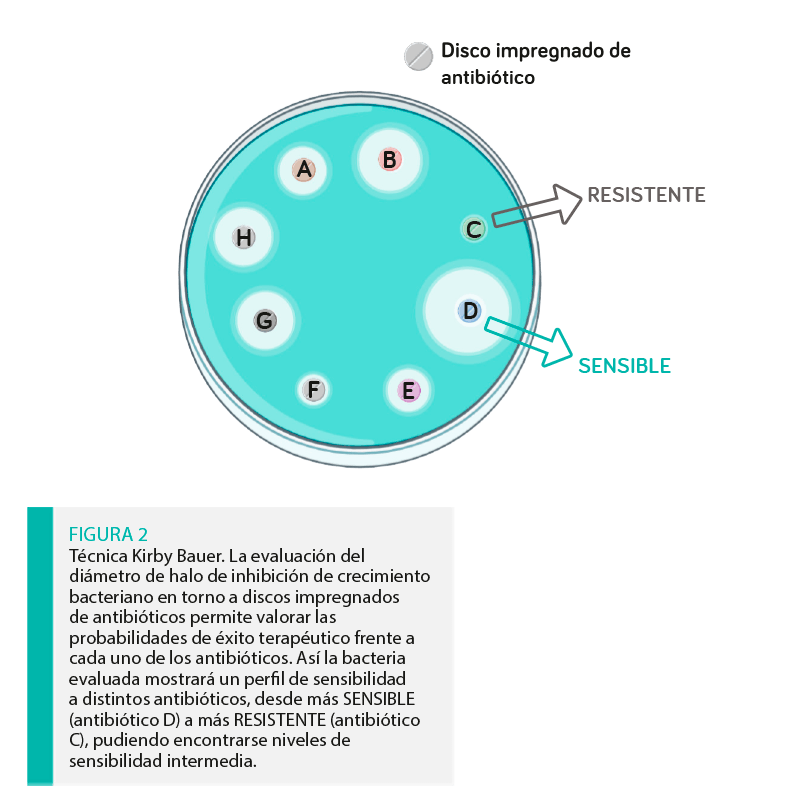
La técnica de Kirby Bauer o disco-placa consiste en sembrar la bacteria en una placa de agar y colocar discos impregnados con una cantidad de antibiótico estandarizada. Si el antibiótico inhibe el crecimiento de la misma, se creará un halo de inhibición de un determinado diámetro. Este diámetro se compara con unos puntos de corte clínicos establecidos previamente determinando si la bacteria es sensible o resistente a cada uno de los antibióticos evaluados (Figura 2).
CONCENTRACIÓN MÍNIMA INHIBITORIA
En la técnica de CMI, la bacteria se enfrenta a diferentes concentraciones de un antibiótico, siendo el valor de concentración mínima inhibitoria la menor concentración frente a la cual la bacteria no es capaz de crecer. Al igual que con la técnica Kirby Bauer, esta concentración se compara con puntos de corte clínicos establecidos para conocer el perfil de sensibilidad. La determinación de la CMI puede realizarse por diferentes métodos, siendo el método de microdilución (Figura 3) y tiras de celulosa los más utilizados.
TÉCNICAS INMUNOLÓGICAS – SEROLOGÍA
Las técnicas inmunológicas permiten evaluar la presencia de inmunoglobulinas (anticuerpos) en suero como respuesta inmunitaria humoral a un agente infeccioso. Se basan en la unión irreversible entre una sustancia antigénica (Ag) como virus, bacteria o parásito y su correspondiente anticuerpo (Ac). La formación de los complejos Ag-Ac se evalúa mediante:
 Marcadores enzimáticos, fluorescentes o quimioluminiscentes.
Marcadores enzimáticos, fluorescentes o quimioluminiscentes.
 Observación de la aglutinación.
Observación de la aglutinación.
EN LA ESPECIE PORCINA LAS TÉCNICAS MÁS UTILIZADAS SON LAS TÉCNICAS INMUNOENZIMÁTICAS (ELISA, PRINCIPALMENTE) Y LAS TÉCNICAS DE AGLUTINACIÓN
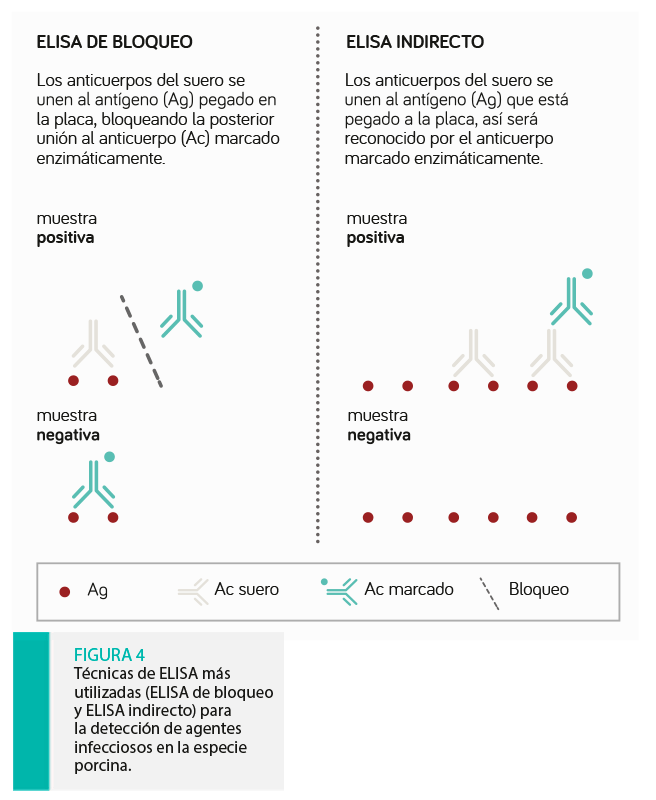
El ELISA (enzyme linked immunosorbent assay) es un tipo de inmunoensayo en el que el Ag se encuentra ligado a una fase sólida (placas de 96 pocillos). Los diseños de los ensayos son diversos (Figura 4) pero todos ellos tienen en común la utilización de enzimas como marcador.
A la hora de seleccionar una técnica de ELISA deberemos valorar una serie de premisas:
 Tipo de inmunoglobulina: se pueden detectar inmunoglobulinas totales, IgM y/o IgG. Tipo de inmunoglobulina: se pueden detectar inmunoglobulinas totales, IgM y/o IgG.
 Las IgM se producen durante las primeras fases de la respuesta inmunitaria. Las IgM se producen durante las primeras fases de la respuesta inmunitaria.
 Las IgG predominan en la respuesta inmunitaria secundaria. Las IgG predominan en la respuesta inmunitaria secundaria.
LA DETECCIÓN EN PARALELO DE IgM E IgG APORTA INFORMACIÓN SOBRE EL TIEMPO DE EVOLUCIÓN DEL PROCESO
 Estado de vacunación de los animales: la mayoría de las técnicas serológicas no diferencian entre anticuerpos vacunales y anticuerpos producidos por una infección, siendo una limitación en caso de animales vacunados. No obstante, existen en el mercado algunos ensayos DIVA (como Aujeszky gE) que permiten la diferenciación si los animales han sido vacunados con vacunas marcadas. Estado de vacunación de los animales: la mayoría de las técnicas serológicas no diferencian entre anticuerpos vacunales y anticuerpos producidos por una infección, siendo una limitación en caso de animales vacunados. No obstante, existen en el mercado algunos ensayos DIVA (como Aujeszky gE) que permiten la diferenciación si los animales han sido vacunados con vacunas marcadas.
 Objetivo del análisis: Objetivo del análisis:
 Los ELISA indirectos están diseñados para evaluar prevalencias de enfermedades a nivel de población, muy útiles en el seguimiento y monitorización de explotaciones positivas. Los ELISA indirectos están diseñados para evaluar prevalencias de enfermedades a nivel de población, muy útiles en el seguimiento y monitorización de explotaciones positivas.
 Los ELISA de bloqueo tienen por objetivo la realización de diagnósticos individuales, siendo habitual su uso para confirmar resultados de otra técnica screening. Los ELISA de bloqueo tienen por objetivo la realización de diagnósticos individuales, siendo habitual su uso para confirmar resultados de otra técnica screening.
Por último, indicar que en ocasiones no es suficiente un único análisis serológico para llegar a una conclusión diagnóstica, sino que se deben realizar dos análisis con una separación de 2-3 semanas para demostrar seroconversión frente al agente infeccioso. |
 TÉCNICA DE AGLUTINACIÓN
TÉCNICA DE AGLUTINACIÓN
La macroaglutinación consiste en la observación macroscópica de la aglutinación por la unión Ag-Ac.
El ejemplo más conocido es la técnica de Rosa de Bengala para la detección de Brucella spp. (Figura 5). Esta técnica es económica y sensible, pero, debido a la reacción cruzada con otras bacterias (Yersinia enterocolitica o algunos serovares de Escherichia coli), los resultados positivos deben ser confirmados.
En el caso de las técnicas de microaglutinación (MAT) es necesaria la utilización de un microscopio para evaluar la aglutinación.
La serología de Leptospira se realiza mediante MAT y es específica de cada serovar. En porcino los serovares más estudiados son L. bratislava y L. pomona al ser los considerados adaptados a la especie.
Las técnicas moleculares son aquellas técnicas basadas en la detección de ácidos nucleicos (ADN o ARN) para determinar la presencia del agente etiológico en la muestra clínica. También se engloban dentro de este grupo aquellas que analizan las proteínas.
En los últimos años, las técnicas moleculares han ido cobrando gran importancia por su rapidez, sensibilidad y especificidad. En comparación al cultivo microbiológico son técnicas mucho más rápidas, obteniéndose resultados en unas pocas horas, y permiten detectar bacterias de difícil crecimiento o virus, cuyo diagnóstico por cultivo resulta mucho más complejo.
Dentro de las técnicas moleculares cabe destacar la PCR a tiempo real, y otras como la secuenciación, microarrays o secuenciación masiva (más conocida como NGS, Next generation sequencing).
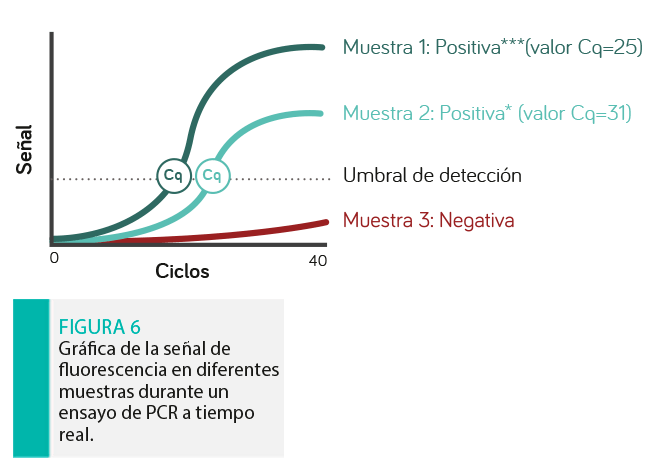
La PCR a tiempo real, también conocida como PCR cuantitativa (qPCR), es la técnica molecular más ampliamente utilizada hoy en día. Consiste en la detección y amplificación de regiones específicas del genoma de los diferentes agentes patógenos.
La detección se realiza tras cada ciclo de PCR gracias al uso de oligonucleótidos marcados con moléculas fluorescentes que se unen a la región amplificada de modo que, a mayor cantidad de genoma del agente, mayor fluorescencia.
El ciclo en el que esta señal supera umbral de detección es lo que se conoce como el valor Cq, de modo que valores Cq menores indican una mayor cantidad del agente en la muestra inicial. Así, utilizando una muestra de ácido nucleico cuantificado se puede determinar la cantidad de patógeno en la muestra inicial.
LOS VALORES CQ MENORES INDICAN UNA MAYOR CANTIDAD DEL AGENTE EN LA MUESTRA INICIAL
La PCR a tiempo real permite detectar, además de bacterias, virus o parásitos, genes codificantes de factores de virulencia, como toxinas, fimbrias, adhesinas, etc. que, en algunas ocasiones, son imprescindibles para evaluar la implicación del agente infeccioso en la patología (por ejemplo, en caso de Escherichia coli o Clostridium perfringens). Esta técnica también permite una identificación a nivel de subespecie (por ejemplo, diferentes serotipos de Streptococcus suis o Actinobacillus pleuropneumoniae).
Entre las técnicas de secuenciación, la más empleada es la secuenciación de Sanger que permite determinar la secuencia de nucleótidos de una o varias regiones específicas del genoma de un determinado patógeno.
Dentro del ámbito de las enfermedades infecciosas, esta herramienta posibilita, entre otros, establecer el origen de un brote, es decir, conocer si la cepa circulante estuvo antes, si se trata de una nueva o si por el contrario dicha cepa es una cepa vacunal.
VARIAS SECUENCIAS PUEDEN REPRESENTARSE EN FORMA DE ÁRBOL FILOGENÉTICO PARA VER LAS RELACIONES ENTRE DIFERENTES CEPAS
La secuenciación es una técnica muy útil para el control y monitorización de ciertas enfermedades como, por ejemplo, el virus del Síndrome Respiratorio y Reproductivo Porcino (PRRS). No obstante, las aplicaciones de la secuenciación pueden ir más allá, por ejemplo:
 Es posible genotipar el rotavirus A en base a las secuencias codificantes de las proteínas virales VP7 (determina el genotipo G) y VP4 (genotipo P).
Es posible genotipar el rotavirus A en base a las secuencias codificantes de las proteínas virales VP7 (determina el genotipo G) y VP4 (genotipo P).
 Se puede genotipar el virus de la Influenza A, cuyo genotipo está definido por las secuencias de la hemaglutinina (H) y la neuraminidasa (N) (ejemplo: H1N1).
Se puede genotipar el virus de la Influenza A, cuyo genotipo está definido por las secuencias de la hemaglutinina (H) y la neuraminidasa (N) (ejemplo: H1N1).
Otro técnica basada en secuenciación que puede ser empleada para el tipado a nivel de subespecie es la de MLST (Multi Locus Sequence Typing), la cual permite clasificar las cepas bacterianas en un tipo u otro (denominado sequence type o ST) en función de la secuencia de ADN de siete de sus genes de acuerdo a una clasificación previamente descrita. Un ejemplo en porcino es la realización de MLST para diferenciar entre cepas de Brachyspira hyodysenteriae.
Por último, cabe destacar las tecnologías de secuenciación de nueva generación (Next Generation Sequencing, NGS) que han aumentado la capacidad de secuenciación, permitiendo secuenciar desde genomas completos a ecosistemas (microbioma, metagenómica) y proporcionando una gran cantidad de información de una sola muestra.
| En definitiva, la base del diagnóstico en el laboratorio comienza en la propia toma de muestras y continúa con la elección de la técnica más adecuada para realizar un correcto diagnóstico que abarca diferentes objetivos, desde la identificación del agente etiológico hasta estudios de tipado o variabilidad de cepas. Es importante tener claro que cada una de las técnicas proporciona un tipo de información diferente y que se pueden complementar aumentando la sensibilidad diagnóstica y permitiendo conocer el agente etiológico de forma precisa. |
[/registrados]








 TÉCNICA DE AGLUTINACIÓN
TÉCNICA DE AGLUTINACIÓN