Norovirus porcino: de patógeno en el olvido a potencial agente zoonótico
Para leer más contenidos de Revista porciSapiens Abril 2024
Para leer más contenidos de Revista porciSapiens Abril 2024
| Los Norovirus son un género de la familia Caliciciviridae que se caracterizan por tener un RNA no segmentado y un virión carente de envoltura. Este patógeno es la principal causa de gastroenteritis aguda de origen no bacteriano en humanos a nivel mundial, y se les describe infectando también a un gran número de especies de animales domésticos y silvestres1. |
La primera descripción de este virus fue en 1972 por Kapikian y col., quienes por microscopia electrónica identificaron partículas virales en muestras procedentes de un brote de gastroenteritis aguda en humanos en Norwalk (Estados Unidos), ciudad que daría lugar al nombre del agente2.
EN EL PORCINO EL PRIMER REPORTE DE NOROVIRUS LO DIERON SUGIEDA Y COL. EN 1997, QUIENES IDENTIFICAN ESTE VIRUS EN HECES DE ANIMALES ORIGINARIOS DE JAPÓN3
Aunque la presencia de norovirus se describe también en perros, gatos, ovinos y bovinos, es en el ganado porcino donde genera especial interés, por la estrecha relación genética entre las cepas circulantes en esta especie animal y las que afectan al humano.
| Desde una perspectiva One Health, el estudio de las infecciones por norovirus porcino adquiere relevancia, dada la posibilidad de esta especie para constituirse en reservorio de cepas con potencial zoonótico. |
DIVERSIDAD GENÉTICA DE LOS NOROVIRUS
El genoma de los norovirus posee tres marcos abiertos de lectura (Open Reading Frames, ORFs).
LA VP1 ES LA PRINCIPAL PROTEÍNA DE LA CÁPSIDE Y DE PARTICULAR IMPORTANCIA PARA CLASIFICAR LA DIVERSIDAD ANTIGÉNICA DE LOS MIEMBROS DE ESTA FAMILIA VIRAL1
Inicialmente, los norovirus fueron clasificados en genogrupos y genotipos en base a las secuencias nucleotídicas del gen RdRp, una polimerasa codificada en el ORF1.
Posteriormente, la secuenciación del gen VP1 demostró ser más fiable para clasificar los genotipos.
Actualmente, se ha propuesto un sistema de clasificación dual, en el que:
|
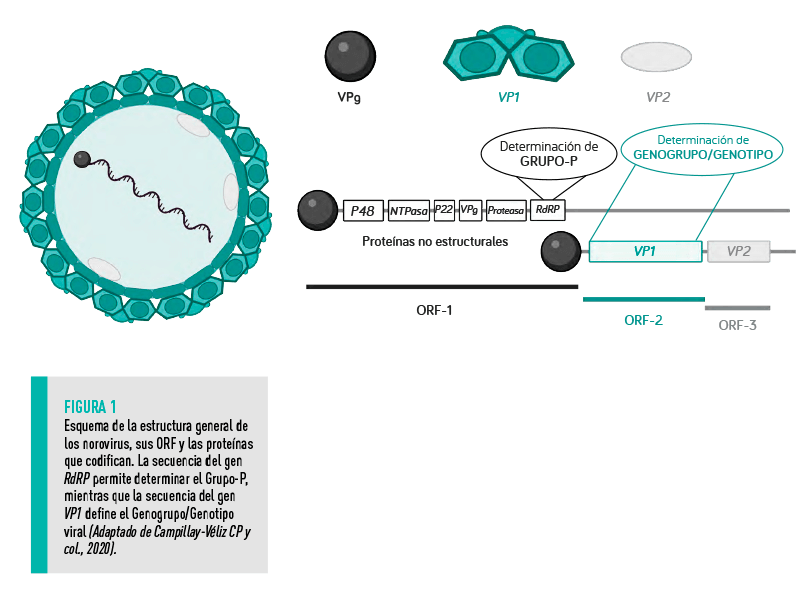
Hasta la fecha, se han descrito un total de 10 genogrupos y 49 genotipos distintos de norovirus, observándose que estos pueden infectar diferentes especies animales dependiendo del genogrupo:[registrados]
| Norovirus en humanos En humanos, el genogrupo GII se ha considerado el más patogénico, con un total de 27 genotipos diferentes según su VP1, siendo el genotipo GII.4 el más prevalente y el que tiene mayores tasas de evolución y mutación5. |
Norovirus en porcinos En el caso del porcino, los estudios indican que los genotipos GII.11, GII.18 y GII.19 son específicos de esta especie animal. Sin embargo, en el año 2007 una cepa humana GII.4 fue descrita en muestras de porcinos en Canadá y posteriormente en Japón en 2010 y en Taiwán en 2012, generando la preocupación sobre el rol de esta especie como reservorio de norovirus zoonóticos1. |

ESTUDIOS SOBRE EL NOROVIRUS PORCINO
La gran diversidad genética y el hecho de no poder ser cultivados in vitro han limitado el estudio de los norovirus durante mucho tiempo.
Aunque la información sobre este agente en humanos ha incrementado considerablemente en los últimos años, sobre todo a nivel molecular, las investigaciones en el porcino son aún escasas, describiéndose recientemente que, hasta el año 2022, solo 47 artículos a nivel global evaluaban la presencia de norovirus en esta especie animal1.
Tras la primera descripción del Norovirus porcino en Japón, este agente se ha descrito en países de los cinco continentes.

Además de los genotipos de norovirus específicos del porcino GII.11, GII.18 y GII.19, y del genotipo humano GII.4 descrito previamente, estudios recientes han identificado la circulación de forma puntual en el porcino de otros genotipos humanos como:
|
 |
Estos hallazgos han dado forma a la hipótesis de que los norovirus no están necesariamente restringidos a infectar un solo tipo de hospedador y que podrían ser capaces de saltar la barrera entre especies1,6. |
No obstante, dos estudios experimentales usando aislados de norovirus porcino han demostrado que este agente puede ocasionar diarrea ligera-moderada con un periodo relativamente prolongado de excreción viral1,7.
IDENTIFICACIÓN Y CARACTERIZACIÓN MOLECULAR DE NOROVIRUS PORCINO EN ESPAÑA
En Europa, solo algunos países han evaluado la presencia de Norovirus porcino en su población ganadera, habiéndose identificado este agente en estudios realizados en Italia, Holanda, Alemania, Eslovenia, Hungría y Bélgica.
En España, solo se ha descrito un estudio (Halalei y col., 2010) en el que evaluaron la presencia de Rotavirus y Calicivirus en 221 muestras fecales de porcino de diferentes granjas en la comunidad autónoma de Aragón. En este caso, aunque detectaron Rotavirus, no lograron identificar casos positivos a norovirus1,8.
| Con el fin de mejorar la información epidemiológica sobre Norovirus en el sector porcino español, se realizó un estudio para identificar y caracterizar molecularmente a este agente en un total de 480 muestras clínicas de procesos digestivos recibidos en nuestro laboratorio (Exopol S.L., Zaragoza) durante el periodo 2020 al 2022. |
MUESTREO
Las muestras consistían en:
|
 |
Fueron analizadas en pools (67%) o de forma individual (33%). |

Adicionalmente, procedían de dos sistemas de producción diferentes:
ANÁLISIS LABORATORIAL
Para la identificación de Norovirus porcino se evaluaron dos ensayos de transcripción reversa con PCR en tiempo real (RT-qPCR) que usaban como diana una región muy conservada entre el ORF1 y el ORF2:
1. Uno era un ensayo previamente descrito por Da Silva y col. (2007)9.
2. El otro fue un ensayo de diseño propio.
La caracterización molecular fue realizada por secuenciación Sanger utilizando diversos protocolos previamente descritos para la amplificación de los genes RdRp y VP110,11,12, así como unos cebadores de diseño propio.
RESULTADOS
IDENTIFICACIÓN MOLECULAR DE NOROVIRUS
![]()
En una evaluación inicial de 74 casos clínicos utilizando ambos ensayos de RT-qPCR, el ensayo de Da Silva y col. identificó como positivos un 5% (4/74) de las muestras, mientras que el diseño propio detectó un 14% (10/74) de positivos.
Adicionalmente, se observó que las muestras positivas con el nuevo diseño tenían un ciclo de cuantificación (Cq) ligeramente inferior que el ensayo de Da Silva y col., por lo que fue utilizado como método de detección en el resto de muestras clínicas.
El análisis posterior del panel total de 480 muestras clínicas demostró que el 11% (52/480) resultaron positivas a Norovirus porcino mediante RT-qPCR, con rangos de Cq entre 25 y 38.

EL NOROVIRUS SE IDENTIFICÓ EN MUESTRAS DE HECES (14%), PERO TAMBIÉN EN TEJIDO INTESTINAL (12%) E HISOPOS (9%)
| La frecuencia de infección por norovirus en nuestro estudio fue muy similar a la de un estudio reciente en granjas de porcino en la región del Véneto en Italia (11%) y a otro estudio realizado por un laboratorio en Bélgica (11%) en el año 200713,14. |

INFLUENCIA DEL SISTEMA PRODUCTIVO
![]()


INFLUENCIA DE LA FASE PRODUCTIVA
![]()
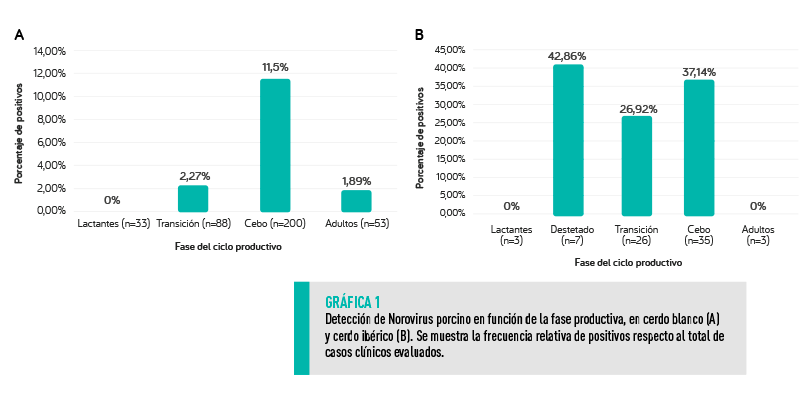
| CERDO BLANCO El análisis de la infección por norovirus según la fase del ciclo productivo demostró que, en el caso de cerdo blanco la mayoría de animales positivos pertenecían a la fase de cebo o engorde (Gráfica 1). Estos resultados son concordantes con estudios previos indicando que, en sistemas de producción intensiva, la mayor frecuencia de infección por norovirus se observa en estas etapas1,13. |
CERDO IBÉRICO En el caso del cerdo ibérico, la frecuencia de positivos en las fases de destete, transición y cebo no presentaron diferencia estadística significativa (Gráfica 1) y no se pudo identificar a este agente en animales lactantes ni adultos. |
DISTRIBUCIÓN GEOGRÁFICA
![]()
Norovirus porcino fue identificado en la mayoría de las regiones evaluadas.
La mayor frecuencia de detección se observó en la Comunidad autónoma de Extremadura con un 37% (22/60), seguido de la Comunidad foral de Navarra con 22% (2/9), Andalucía con 10% (2/21), Comunidad Valencia con 8% (2/26), Aragón con 7% (16/214), Castilla y León con 7% (3/44) y Cataluña con 6% (4/68).
COINFECCIONES
![]()
Es interesante resaltar que, en más de la mitad de las muestras positivas a norovirus (27/52), este agente estuvo en coinfección con uno o más patógenos digestivos, principalmente Brachyspira hyodysenteriae y Lawsonia intracellularis. No obstante, en un 13% (7/52) de las muestras positivas, Norovirus porcino fue el único agente identificado.
ESTOS HALLAZGOS SUBRAYAN LA NECESIDAD DE REALIZAR ESTUDIOS ADICIONALES PARA DETERMINAR EL VERDADERO ROL DE ESTE AGENTE EN LOS PROBLEMAS DIGESTIVOS DEL PORCINO
CARACTERIZACIÓN MOLECULAR Y ANÁLISIS FILOGENÉTICO
![]()
A pesar del uso de distintos protocolos de secuenciación, solo fue posible obtener secuencias parciales de la región de unión del gen RdRP y VP1 en un total de siete muestras procedentes de cinco provincias distintas.
El uso de la herramienta bioinformática Norovirus Typing Tool vs.2.0 permitió el genotipado de las cepas como GII.11 y GII.18. Esta última pudo ser asignada como GII.P18.

En el análisis filogenético de las secuencias de norovirus de este estudio se incluyeron secuencias de referencia de:
Los resultados del análisis filogenético distinguieron dos grupos bien diferenciados (Figura 2).
| CLADO A El clado A contenía las secuencias de:
Dentro de los genotipos específicos del porcino, el GII.18 se describe como el más relacionado genéticamente con los norovirus humanos, generando preocupación por su potencial peligro para la salud humana1. |
CLADO B En el clado B se observaron dos subgrupos:
Cabe resaltar que el análisis filogenético de las cepas de nuestro estudio confirmó la inclusión de estas muestras como GII.18 y GII.11, validando la identificación previa de estas secuencias mediante la herramienta online de genotipado. |
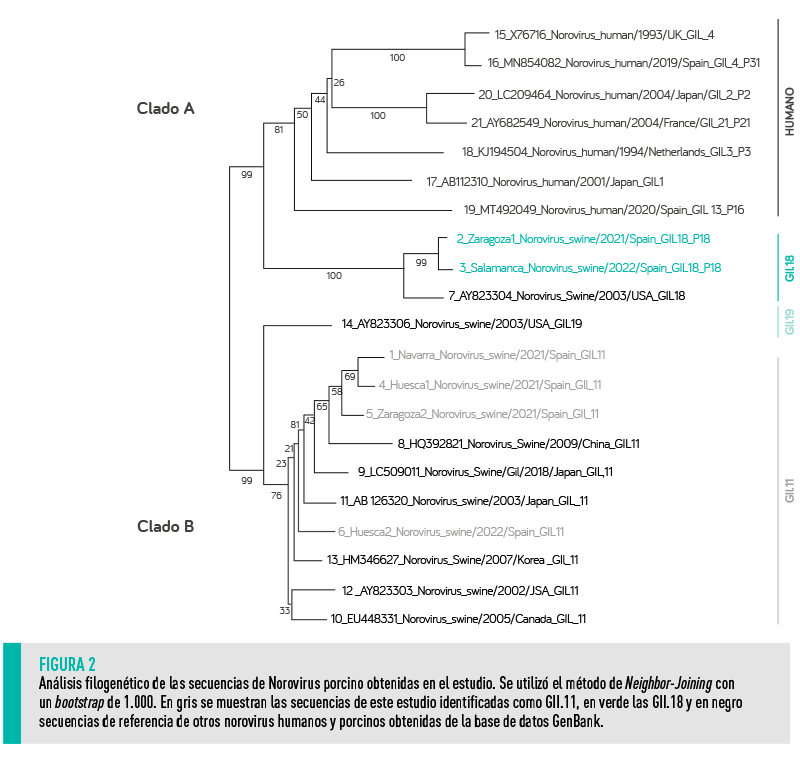
Las cepas de norovirus de este estudio presentaron una identidad nucleotídica mayor al 93% con varias secuencias de norovirus porcino depositadas en el GenBank. Sin embargo, entre ellas el porcentaje de identidad nucleotídica estuvo entre un 79% y 98%, reflejando la diferencia entre los dos genotipos de norovirus porcino identificados.
LA IDENTIDAD NUCLEOTÍDICA ENTRE LAS CEPAS GII.11 ESTUVO ENTRE UN 93% Y 97%, MIENTRAS QUE ENTRE LAS DOS GII.18 FUE DEL 98%
CONCLUSIÓN
Este estudio es el primer reporte de la identificación molecular de Norovirus porcino en casos clínicos de procesos digestivos en nuestro país. Asimismo, se describe la circulación de los genotipos GII.11 y GII.18 en varias provincias del país. Aunque no fue posible identificar genotipos de norovirus potencialmente zoonótico, es importante continuar la investigación sobre este agente, tanto por su impacto clínico en la especie porcina como desde la perspectiva One Health, con el fin de minimizar los riesgos que puede suponer para el ser humano.
BIBLIOGRAFÍA
1. Cavicchio L y col. Swine Norovirus: Past, Present, and Future. Viruses. 2022 Mar 5;14(3):537
2. Lucero Y, Matson DO, Ashkenazi S, George S, O’Ryan M. Norovirus: Facts and Reflections from Past, Present, and Future. Viruses. 2021 Nov 30;13(12):2399
3. Sugieda M y col. Detection of Norwalk-like virus genes in the caecum contents of pigs. Arch Virol. 1998;143(6):1215-21.
4. Chhabra P y col. Updated classification of norovirus genogroups and genotypes. J Gen Virol. 2019 Oct;100(10):1393-1406.
5. Koo ES, Jeong YS. Comparative study of the geographical spread of genogroup II porcine norovirus and human norovirus. J Microbiol. 2021 Jul; 59(7):644-650
6. Villabruna N, Koopmans MPG, de Graaf M. Animals as Reservoir for Human Norovirus. Viruses. 2019 May 25;11(5):478
7. Wang QH y col. Porcine noroviruses related to human noroviruses. Emerg Infect Dis. 2005 Dec;11(12):1874-81
8. Halaihel N y col. Enteric calicivirus and rotavirus infections in domestic pigs. Epidemiol Infect. 2010 Apr;138(4):542-8
9. da Silva AK y col. Evaluation of removal of noroviruses during wastewater treatment, using real-time reverse transcription-PCR: different behaviors of genogroups I and II. Appl Environ Microbiol. 2007 Dec;73(24):7891-7
10. Wollants E y col. A decade of norovirus genetic diversity in Belgium. Infect Genet Evol. 2015 Mar; 30:37-44
11. Andrade JSR y col. Detection and molecular characterization of emergent GII.P17/GII.17 Norovirus in Brazil, 2015. Infect Genet Evol. 2017 Jul; 51:28-32
12. Kabue JP y col. Genetic characterisation of Norovirus strains in outpatient children from rural communities of Vhembe district/South Africa, 2014- 2015. J Clin Virol. 2017 Sep; 94:100-106
13. Cavicchio L y col. Unrevealed genetic diversity of GII Norovirus in the swine population of Northeast Italy. Sci Rep. 2020 Jun 8;10(1):9217
14. Mauroy A y col. Noroviruses and sapoviruses in pigs in Belgium. Arch Virol. 2008;153(10):1927-31
15. Campillay-Véliz CP, Carvajal JJ, Avellaneda AM, Escobar D, Covián C, Kalergis AM and Lay MK (2020) Human Norovirus Proteins: Implications in the Replicative Cycle, Pathogenesis, and the Host Immune Response. Front. Immunol. 11:961. doi:10.3389/fimmu.2020.00961
[/registrados]
