Detección y caracterización genética de virus entéricos en brotes de diarrea de explotaciones porcinas en España
Para leer más contenidos de Revista porciSapiens Octubre 2023
Para leer más contenidos de Revista porciSapiens Octubre 2023
| El objetivo de este trabajo fue estudiar la prevalencia y distribución de Astrovirus porcino (PAstV), Kobuvirus porcino (PKoV), Torovirus porcino (PToV), Orthoreovirus de mamíferos (MRV) y Mastadenovirus porcino (PAdV), así como su asociación con virus ampliamente reconocidos como causantes de diarrea en porcino como coronavirus (CoV) y rotavirus (RV) en brotes de diarrea en explotaciones porcinas españolas. Además, se caracterizó genéticamente una selección de las cepas virales. |
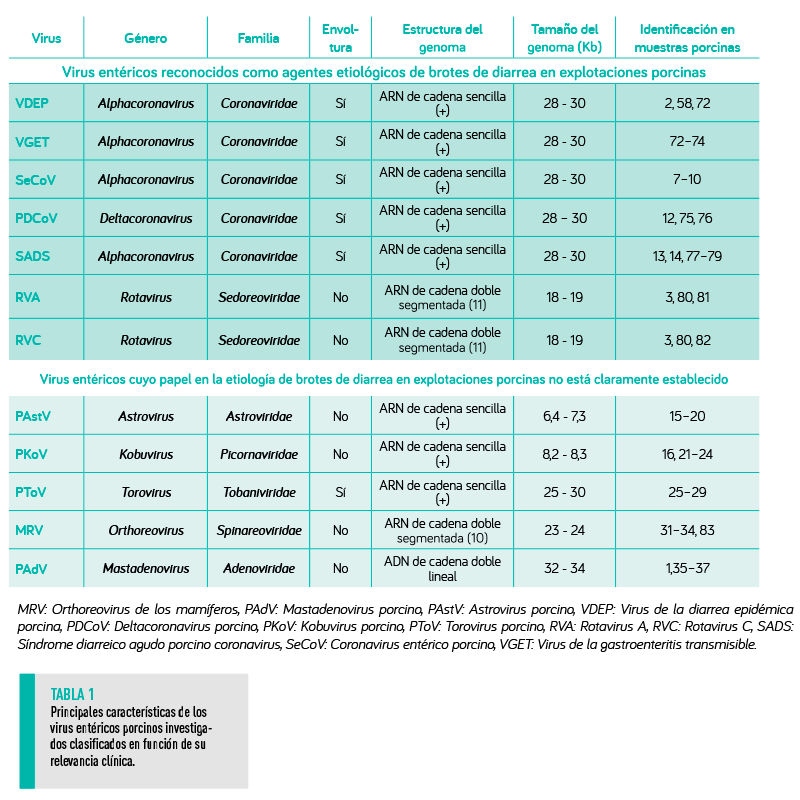
VIRUS ENTÉRICOS PORCINOS
LOS SOSPECHOSOS HABITUALES
Las enfermedades entéricas causadas por virus son muy prevalentes y tienen un impacto negativo en la producción porcina1.
Los coronavirus (CoV) y los rotavirus (RV) son los virus más comunes y reconocidos como responsables de la diarrea en cerdos2-4 (Tabla 1).
ROTAVIRUS
Entre los RV, Rotavirus A (RVA) y Rotavirus C (RVC) son las especies más relevantes3 y están implicados principalmente en brotes de diarrea en lechones lactantes y destetados, aunque pueden detectarse en todas las fases de la producción porcina3.
CORONAVIRUS
Más recientemente, un virus recombinante entre el VGET y el VDEP, conocido como Coronavirus entérico porcino (SeCoV), ha sido identificado en granjas porcinas europeas7-10, causando una enfermedad diarreica idéntica a la causada por el VGET y el VDEP3,11.
Asimismo, otros dos CoV porcinos, el Deltacoronavirus porcino (PDCoV)12 y el Coronavirus del síndrome de diarrea aguda porcina (SADS-CoV)13,14, un Alphacoronavirus, también se han descrito como causa de brotes de diarrea, aunque nunca se han detectado en explotaciones porcinas europeas.
OTROS VIRUS ENTÉRICOS
Además de estos agentes infecciosos, en granjas porcinas se han detectado otros virus entéricos (Tabla 1) en cerdos con diarrea en todo el mundo, aunque su papel etiológico sigue sin estar claro:
|
 |
Estos virus se identifican frecuentemente en coinfecciones con CoV y RV4,38-45, pero también se detectan habitualmente en animales sanos39,46-50. La enteropatogenicidad de PAstV, MRV y PAdV se ha demostrado en lechones convencionales infectados experimentalmente51-55, pero no conocemos la existencia de desafíos experimentales con PKoV o PToV. |
VIRUS ENTÉRICOS PORCINOS EN ESPAÑA

1. Estudiar la prevalencia y distribución de los virus entéricos PAstV, PKoV, PToV, MRV y PAdV en brotes de diarrea en explotaciones porcinas de España.
2. Identificar su asociación con virus ampliamente reconocidos como causantes de diarrea, como CoV y RV.
3. Caracterizar una selección de las cepas virales obtenidas para investigar sus relaciones filogenéticas.
| En conjunto, los resultados obtenidos aportan información sobre la etiología de las enfermedades entéricas en porcino que puede ser utilizada para diseñar e implementar estrategias de diagnóstico y control adecuadas. |
MATERIAL Y MÉTODOS
RECOGIDA Y PROCESAMIENTO DE LAS MUESTRAS
![]()
El estudio se llevó a cabo [registrados]entre enero de 2017 y octubre de 2020 en 206 explotaciones porcinas españolas con brotes de diarrea que afectaban a:
De cada granja se recibieron 2-6 muestras de heces individuales y el veterinario responsable de la explotación clasificó el brote, en función de criterios clínicos como la velocidad de progresión de la enfermedad o la respuesta a los antimicrobianos, como:
Las muestras individuales se mezclaron para preparar una muestra conjunta de heces por granja que se diluyó 1:2 (v/v) en solución salina estéril tamponada con fosfato (PBS), se homogeneizó en vórtex y se clarificó mediante centrifugación.
El ácido nucleico se extrajo a partir de 140 μl del sobrenadante y utilizando un kit comercial (QIAMP Viral RNA and DNA Mini Kit, QIAGEN), siguiendo las instrucciones del fabricante. Tras la extracción se almacenó a -80 °C hasta su uso.
DETECCIÓN MOLECULAR DE VIRUS ENTÉRICOS PORCINOS
![]()
Se llevaron a cabo cuatro PCRs de transcripción reversa o RT-PCR múltiplex utilizando el kit Verso 1-Step RT-PCR ReddyMix (Thermo Scientific) para la detección de:
Las reacciones se llevaron a cabo en las siguientes condiciones: 50 °C durante 30 min, 95 °C durante 2 min, 45 ciclos a 95 °C durante 20 s, 50 °C durante 30 s y 72 °C durante 1 min y 30 s, seguidos de un paso final de extensión a 72 °C durante 10 min.

SECUENCIACIÓN Y ANÁLISIS FILOGENÉTICO
![]()
Se seleccionaron 14 muestras para su secuenciación en función de los resultados de la RT-PCR (muestras con el mayor número de virus diferentes detectados).

Finalmente, las secuencias obtenidas fueron alineadas y comparadas filogenéticamente con otras secuencias completas disponibles en la plataforma GenBank.

ANÁLISIS ESTADÍSTICO
![]()
Los análisis estadísticos de los datos se realizaron mediante las pruebas exacta y ANOVA de Fisher utilizando Epi InfoTM para un nivel de confianza del 95%.
RESULTADOS
PREVALENCIA Y DISTRIBUCIÓN DE LOS DISTINTOS VIRUS ENTÉRICOS PORCINOS EN LOS BROTES DE DIARREA EN ESPAÑA
![]()
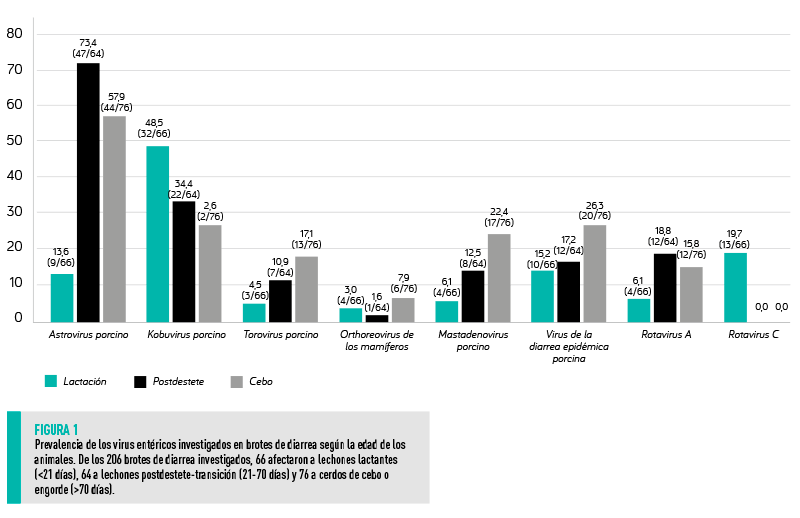
Se detectó al menos uno de los virus entéricos investigados en 160 de los 206 brotes investigados (77,7%).
PAstV fue el virus más frecuentemente detectado (48,5%; n = 100), seguido de PKoV (27,2%; n = 56), VDEP (19,9%; n = 41), PAdV (14,1%; n = 29), RVA (13,6%; n = 28), PToV (11,2%; n = 23), RVC (6,3%; n = 13) y MRV (4,4 %; n = 9).
NO SE DETECTARON EN NINGUNA DE LAS EXPLOTACIONES INVESTIGADAS EL VGET NI EL SeCoV
La prevalencia de brotes de diarrea positivos (un resultado positivo para al menos uno de los virus analizados) fue similar (p = 0,787) entre los periodos de lactación (75,8%, 50 de 66), postdestete-transición (76,6%, 49 de 64) y engorde (80,3%, 61 de 76). Sin embargo, se observaron diferentes patrones de distribución por edades para cada uno de los virus investigados (Figura 1).
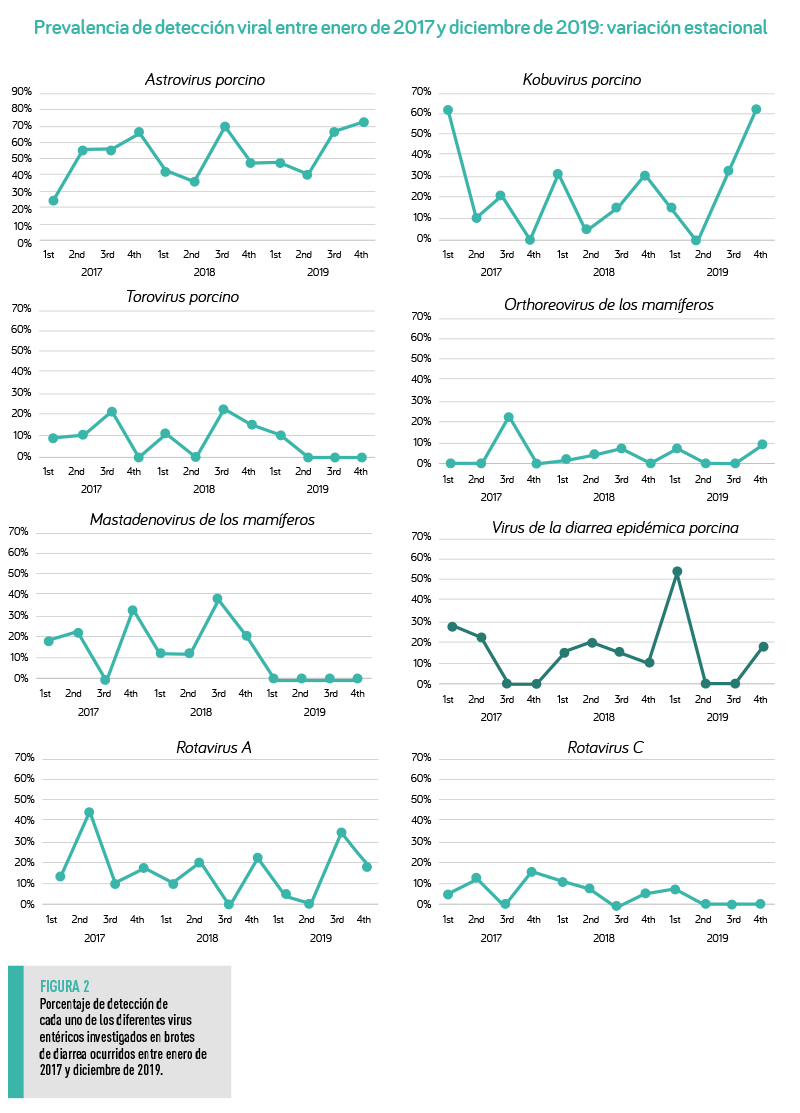
LA MAYORÍA DE LOS BROTES INVESTIGADOS (50,0%; 103 DE 206) SE PRODUJERON DURANTE EL PRIMER TRIMESTRE EN COMPARACIÓN CON EL RESTO DEL AÑO (P < 0,001)
La distribución estacional fue clara para el VDEP que se detectó con mayor frecuencia durante el invierno (de enero a marzo) (p = 0,011), pero no se observó para ninguno de los otros virus entéricos investigados (Figura 2).
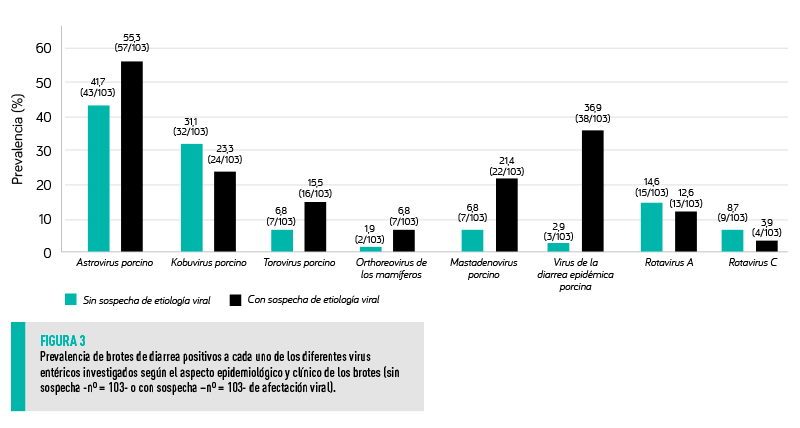
El riesgo de resultado positivo a, al menos, uno de los virus analizados fue mayor en los brotes en los que el veterinario clínico indicó que existía sospecha de etiología viral (OR= 2,52; IC 95%: 1,27-5,04) en comparación con los brotes sin sospecha vírica (85,4% frente a 69,9%).

PREVALENCIA Y DISTRIBUCIÓN DE LAS COINFECCIONES VÍRICAS EN LOS BROTES DE DIARREA EN ESPAÑA
![]()
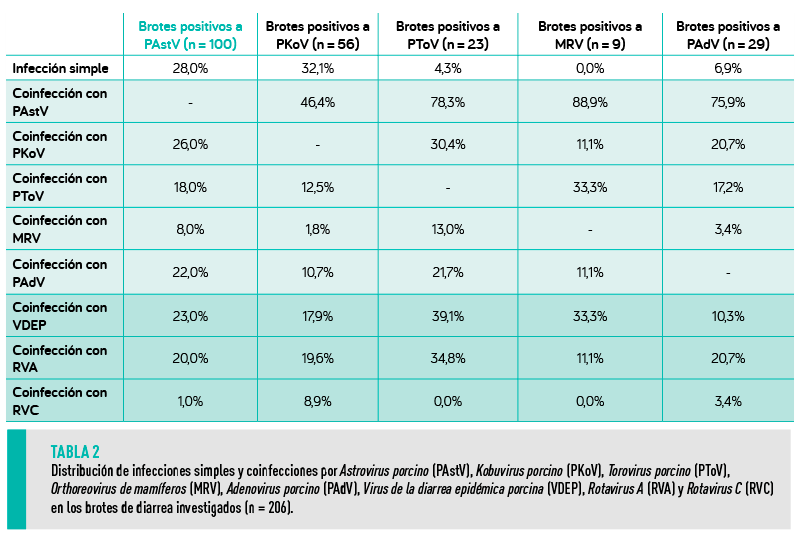
En 69 de los 206 brotes analizados (33,5%) se detectó un único agente viral, mientras que 56 (27,2%) presentaron coinfección con dos virus al mismo tiempo y 35 (17,0%) fueron positivos a más de dos (hasta 5 especies víricas diferentes detectadas simultáneamente en tres granjas distintas).

Tal y como se muestra en la Tabla 2, PAstV y PKoV se detectaron en casi el 30% de los brotes positivos como infección única, mientras que PToV, MRV y PAdV se detectaron en más del 90% de los brotes positivos coinfectando con el VDEP, RV u otros virus entéricos.
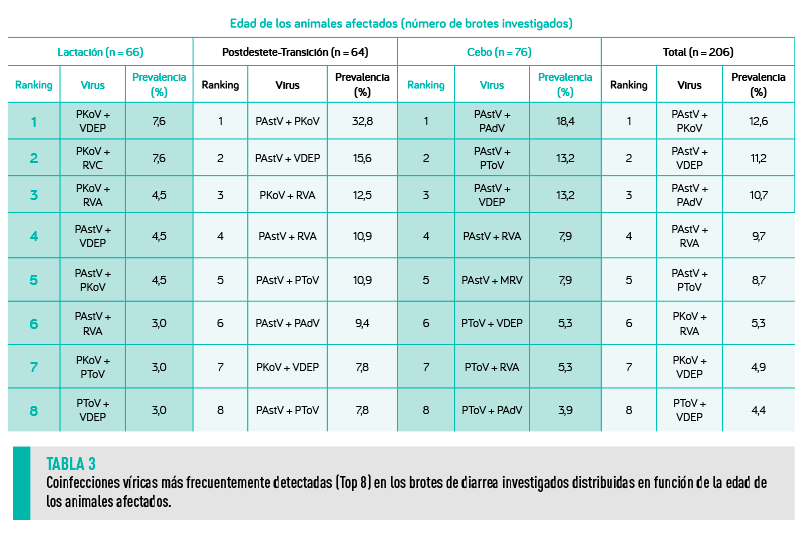
Las combinaciones virales más frecuentes (Top 8) se muestran en la Tabla 3.

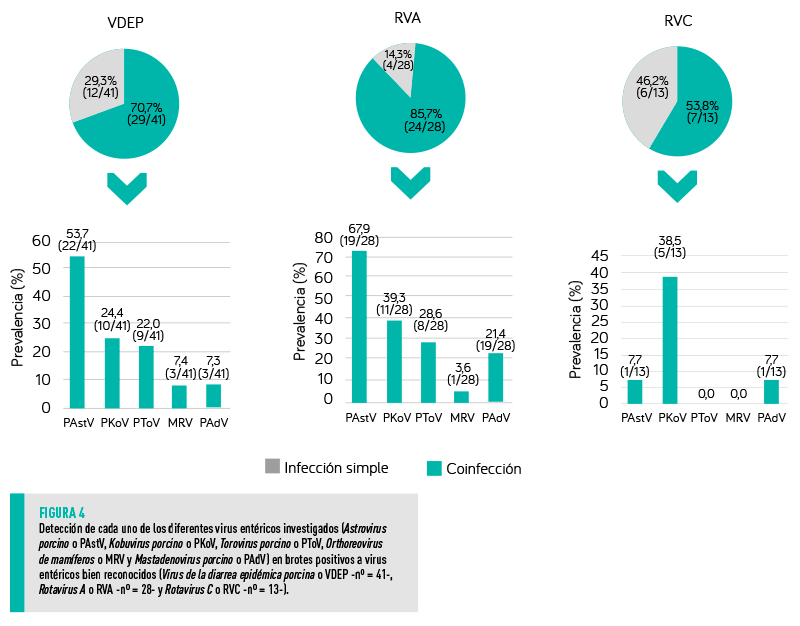
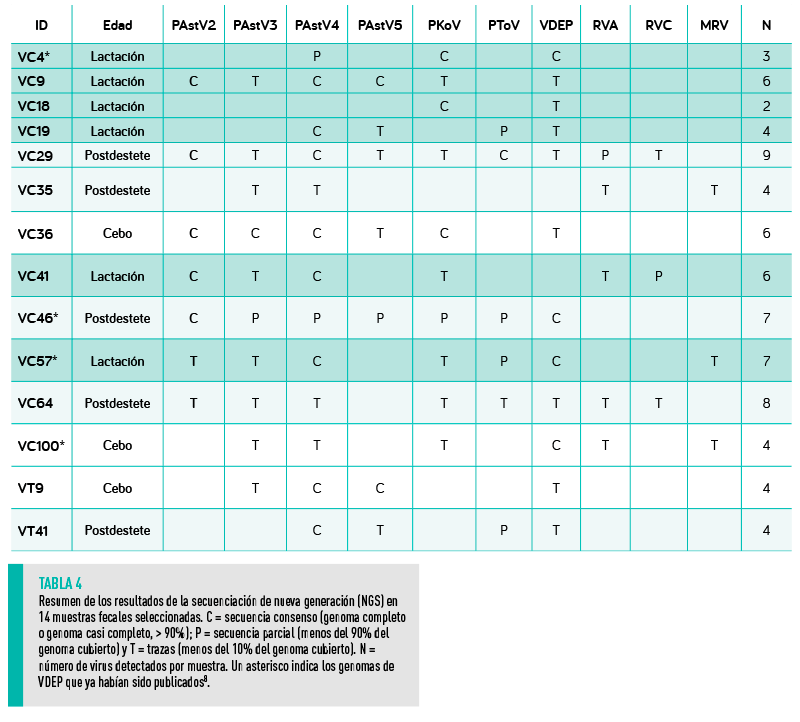
SECUENCIACIÓN Y ANÁLISIS FILOGENÉT ICO DE PAstV, PKoV Y PToV
![]()
Los resultados de la secuenciación de nueva generación (NGS) en 14 muestras de heces se muestran en la Tabla 4.

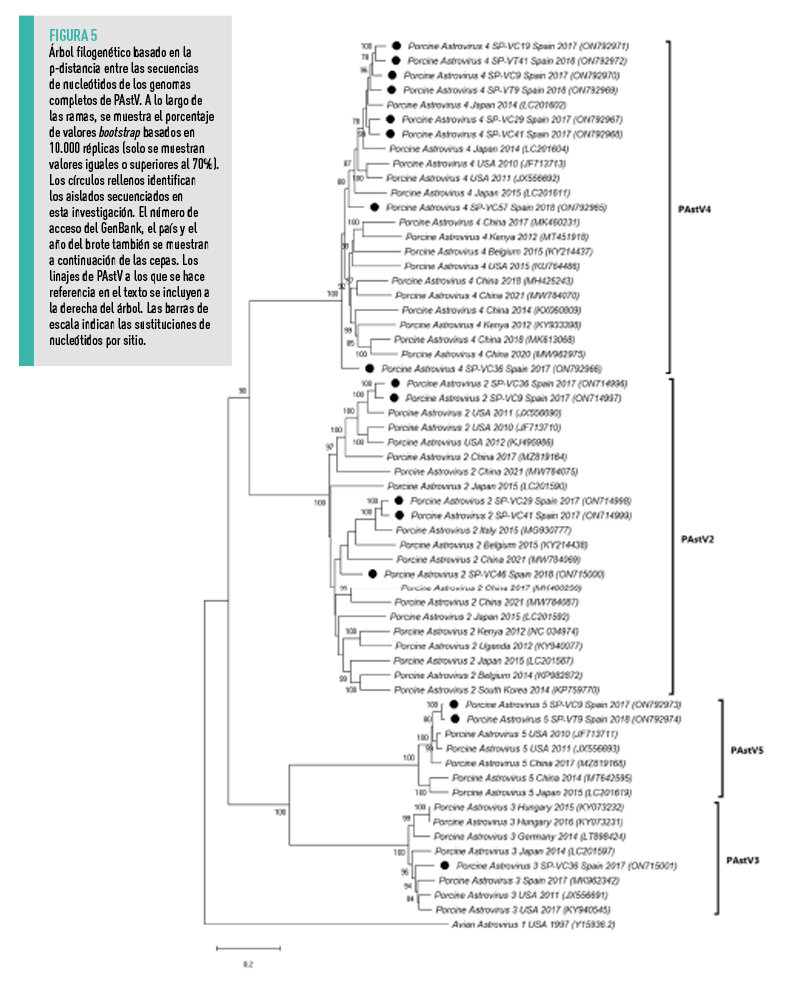
| PAstV SE DETECTARON CUATRO ESPECIES DE PAstV (PAstV2 A PAstV5), SIENDO PAstV4 Y PAstV2 LAS MÁS REPRESENTADAS
La mayoría de las secuencias españolas de PAstV se agruparon con cepas norteamericanas del mismo linaje, aunque PAstV4 se agrupó con secuencias de Japón y algunas de PAstV2 con secuencias de Europa y China (Figura 5). |
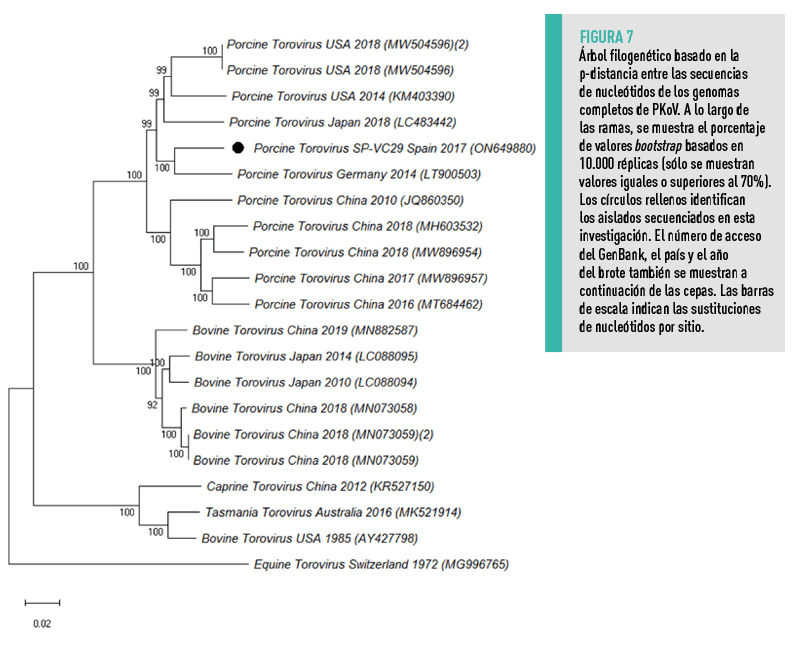
| PKoV En cuanto al análisis filogenético de PKoV (Figura 6), las tres secuencias obtenidas en este estudio se agruparon con otros aislados españoles y húngaros descritos con anterioridad.
|

| PToV Se obtuvo un único genoma completo de PToV y, cuando se comparó con PToV descritos previamente, su identidad nucleotídica fue del 88,6-94,1%, mientras que cuando se comparó con secuencias de Torovirus de otras especies animales fue del 77,3-87,9%.
|

DISCUSIÓN
El papel enteropatógeno de PAstV, PKoV, PToV, MRV y PAdV sigue siendo controvertido y los estudios de casos y controles que han comparado las ratios de detección en cerdos con diarrea y en cerdos sanos no han logrado, por el momento, clarificar esta cuestión39,46-50.
PREVALENCIAS
![]()
En este estudio nos planteamos como objetivo determinar, como punto de partida, la prevalencia de estas especies víricas entéricas en brotes de diarrea de diferentes edades y estudiar su asociación con enteropatógenos víricos bien reconocidos como los CoV (VDEP, VGET y SeCoV) y los RV (RVA y RVC).

NUESTROS RESULTADOS CONFIRMAN QUE LAS INFECCIONES POR PAstV Y PKoV SON ALTAMENTE PREVALENTES EN LAS GRANJAS PORCINAS EUROPEAS, TAL Y COMO HABÍAN DESCRITO VARIOS ESTUDIOS19,46,49,50,56

En concordancia con un informe previo en Hungría49, el PAdV fue menos frecuente (14,1% de los brotes positivos) mientras que el PToV solo se identificó en el 11,2% de las granjas investigadas, en contraposición a la amplia distribución de este virus evidenciada por otro estudio español que estimó más del 99% de animales seropositivos entre los cerdos de más de 11 semanas de edad57.
Por último, el MRV se detectó con menor frecuencia (4,4%), no existiendo datos previos sobre su prevalencia en las explotaciones porcinas europeas.
| En cuanto a los CoV podemos destacar que no se detectaron ni el VGET ni el SeCoV, identificándose el VDEP en el 19,9% de los brotes de diarrea investigados. Finalmente, la prevalencia de brotes positivos a RVA y RVC fue del 13,6 y el 6,3%, respectivamente. |
DISTRIBUCIÓN POR EDADES
![]()
Se observaron tres patrones de distribución por edades entre los virus entéricos investigados.



Estudios previos en granjas porcinas europeas han informado de una tendencia similar para el PAstV46,49,50. Como se ha descrito para los RVA en granjas porcinas1, la disminución de anticuerpos maternales específicos podría explicar el aumento de la prevalencia del PAstV en el periodo postdestete.

ESTACIONALIDAD
![]()
Solo se demostró una distribución estacional para los brotes positivos de VDEP, que fueron más comunes durante los meses más fríos del año (enero-marzo).

NO SE HA DESCRITO ESTACIONALIDAD PARA LOS RV PORCINOS3
SOSPECHA DE ETIOLOGÍA VIRAL
![]()
Curiosamente, los criterios clínicos veterinarios que sugieren una presunta etiología viral se asociaron con la detección viral mediante análisis moleculares.
Esto fue particularmente evidente para los brotes positivos al VDEP, pero también para PAdV y en menor medida para PAstV, PToV o MRV. Por el contrario, RVA, RVC y PKoV se detectaron principalmente en brotes sin sospecha vírica.

COINFECCIONES
![]()
Las coinfecciones fueron relativamente frecuentes, detectándose dos o más virus en casi el 40% de los brotes investigados.

En concordancia con estudios anteriores realizados en Europa49, China4,47 o Canadá44, el número de especies víricas diferentes detectadas en un brote aumenta con la edad de los cerdos afectados.
LAS INFECCIONES SIMPLES FUERON MÁS FRECUENTES EN LECHONES LACTANTES, MIENTRAS QUE EN CERDOS POSTDESTETE Y DE ENGORDE SE OBSERVARON COINFECCIONES SIMULTÁNEAS CON HASTA CINCO VIRUS
Muchos factores asociados al destete, como los cambios en la dieta, la colocación en un entorno nuevo y más contaminado, la mezcla de cerdos o la pérdida de inmunidad pasiva, pueden favorecer la infección por diferentes especies víricas. Además, se ha propuesto que la maduración del sistema inmunitario asociado al intestino tras el destete puede prevenir el desarrollo de enfermedades entéricas asociadas a infecciones simples para aquellos virus entéricos con una enteropatogenicidad moderada64.
SON NECESARIAS MÁS INVESTIGACIONES PARA DESCIFRAR LAS INTERACCIONES ENTRE LOS VIRUS ENTÉRICOS QUE PUEDEN DESEMPEÑAR UN PAPEL PRIMARIO O SECUNDARIO EN LA ETIOLOGÍA DE LA ENFERMEDAD ENTÉRICA EN CERDOS
Cabe señalar que los enfoques cuantitativos basados en la PCR cuantitativa (qPCR) y la secuenciación de nueva generación (NGS), que permiten estimar la carga viral, podrían aportar información adicional relevante en la investigación de la etiología de infecciones entéricas del ganado porcino.
Como proponen Cortey y colaboradores63, la determinación de la abundancia relativa de las distintas especies virales en las coinfecciones permite identificar el virus predominante que, con toda probabilidad, puede proponerse como agente etiológico primario.
ANÁLISIS FILOGENÉTICO
![]()
Por último, el análisis filogenético reveló que la mayoría de las cepas de PAstV se clasificaban como PAstV4 (50,0%, 8/16) y PAstV2 (31,2%, 5/16), siendo estas dos especies las más prevalentes en cerdos con diarrea en España, de forma similar a lo descrito en varios países europeos46,50,67, en Norteamérica68 y en Asia39,69,70.
Los análisis filogenéticos de PKoV revelaron que todas las secuencias completas españolas disponibles (incluidas las tres identificadas en nuestro estudio) estaban incluidas en un único clúster local junto con una cepa húngara y bien diferenciadas de otros aislados europeos38, mientras que la única secuencia completa del genoma de PToV obtenida en esta investigación se agrupó junto con otros aislados de PToV de Europa, América o Asia25,26.

CONCLUSIONES

El hecho de que estos virus entéricos estén frecuentemente implicados en coinfecciones entre ellos y con enteropatógenos virales bien reconocidos, como CoV o RV, hace necesarios estudios adicionales de vigilancia y caracterización, así como investigaciones para evaluar su potencial patogénico tanto en infecciones experimentales como naturales.

Te puede interesar: Diversidad genética de coronavirus porcinos implicados en brotes de diarrea en España
BIBLIOGRAFÍA
1. Thomson JR, Friendship RM. Digestive System. In: Zimmerman JJ, Karriker LA, Ramirez A, Schwartz K, Stevenson GW, Zhang J, editors. Dis swine. 11th ed. Hoboken: John Wiley & Sons; 2019. p. 234–63.
2. Pensaert MB, Martelli P. Porcine epidemic diarrhea: A retrospect from Europe and matters of debate. Virus Res. 2016;226:1–6.
3. Vlasova AN, Amimo JO, Saif LJ. Porcine rotaviruses: Epidemiology, immune responses and control strategies. Viruses. 2017;9:1–27.
4. Zhang Q, Hu R, Tang X, Wu C, He Q, Zhao Z, et al. Occurrence and investigation of enteric viral infections in pigs with diarrhea in China. Arch Virol. 2013;158:1631–6.
5. Wang Q, Vlasova AN, Kenney SP, Saif LJ. Emerging and re-emerging coronaviruses in pigs. Curr Opin Virol. 2019;34:39–49.
6. Jung K, Saif LJ, Wang Q. Porcine epidemic diarrhea virus (VDEP): An update on etiology, transmission, pathogenesis, and prevention and control. Virus Res. 2020;286.
7. Boniotti MB, Papetti A, Lavazza A, Alborali G, Sozzi E, Chiapponi C, et al. Porcine epidemic diarrhea virus and discovery of a recombinant swine enteric coronavirus, Italy. Emerg Infect Dis. 2016;22:83–7.
8. de Nova PJG, Cortey M, Díaz I, Puente H, Rubio P, Martín M, et al. A retrospective study of porcine epidemic diarrhoea virus (VDEP) reveals the presence of swine enteric coronavirus (SeCoV) since 1993 and the recent introduction of a recombinant VDEP-Se-CoV in Spain. Transbound Emerg Dis. 2020;67:2911–2922.
9. Belsham GJ, Rasmussen TB, Normann P, Vaclavek P, Strandbygaard B, Bøtner A. Characterization of a Novel Chimeric Swine Enteric Coronavirus from Diseased Pigs in Central Eastern Europe in 2016. Transbound Emerg Dis. 2016;63:595–601.
10. Akimkin V, Beer M, Blome S, Hanke D, Höper D, Jenckel M, et al. New chimeric porcine coronavirus in Swine Feces, Germany, 2012. Emerg Infect Dis. 2016;22:1314–5.
11. Puente H, Díaz I, Arguello H, Mencía-Ares Ó, Gómez-García M, Pérez-Pérez L, et al. Characterization and cross protection of experimental infections with SeCoV and two VDEP variants. Transbound Emerg Dis. 2022;1–13.
12. Woo PCY, Lau SKP, Lam CSF, Lau CCY, Tsang AKL, Lau JHN, et al. Discovery of Seven Novel Mammalian and Avian Coronaviruses in the Genus Deltacoronavirus Supports Bat Coronaviruses as the Gene Source of Alphacoronavirus and Betacoronavirus and Avian Coronaviruses as the Gene Source of Gammacoronavirus and Deltacoronavi. J Virol. 2012;86:3995–4008.
13. Gong L, Li J, Zhou Q, Xu Z, Chen L, Zhang Y, et al. A new bat-HKU2–like coronavirus in swine, China, 2017. Emerg Infect Dis. 2017;23:1607–9.
14. Pan Y, Tian X, Qin P, Wang B, Zhao P, Yang Y-L, et al. Discovery of a novel swine enteric alphacoronavirus (SeACoV) in southern China. Vet Microbiol. Elsevier; 2017;211:15–21.
15. Bhatta TR, Chamings A, Alexandersen S. Exploring the cause of diarrhoea and poor growth in 8–11-week-old pigs from an australian pig herd using metagenomic sequencing. Viruses. 2021;13.
16. Capai L, Piorkowski G, Maestrini O, Casabianca F, Masse S, de Lamballerie X, et al. Detection of porcine enteric viruses (Kobuvirus, Mamastrovirus and Sapelovirus) in domestic pigs in Corsica, France. PLoS One. 2022;17:1–15.
17. Flores C, Ariyama N, Bennett B, Mena J, Verdugo C, Mor S, et al. Case Report: First Report and Phylogenetic Analysis of Porcine Astroviruses in Chile. Front Vet Sci. 2021;8.
18. Rawal G, Linhares DCL. Scoping review on the epidemiology, diagnostics and clinical significance of porcine astroviruses. Transbound Emerg Dis. 2022;69:974–85.
19. Stamelou E, Giantsis IA, Papageorgiou K V., Petridou E, Davidson I, Polizopoulou ZS, et al. Epidemiology of Astrovirus, Norovirus and Sapovirus in Greek pig farms indicates high prevalence of Mamastrovirus suggesting the potential need for systematic surveillance. Porc Heal Manag. BioMed Central; 2022;8:1–15.
20. Tassoni L, Zamperin G, Schiavon E, Vendramin V, Cavicchio L, Mion M, et al. First whole genome characterization of porcine astrovirus detected in swine faeces in Italy. Vet Ital. 2019;55:221–9.
21. Akagami M, Ito M, Niira K, Kuroda M, Masuda T, Haga K, et al. Complete genome analysis of porcine kobuviruses from the feces of pigs in Japan. Virus Genes. Springer US; 2017;53:593–602.
22. Nantel-Fortier N, Lachapelle V, Letellier A, L’Homme Y, Brassard J. Kobuvirus shedding dynamics in a swine production system and their association with diarrhea. Vet Microbiol. Elsevier; 2019;235:319–26.
23. Milićević V, Kureljušić B, Maksimović-Zorić J, Savić B, Spalević L, Žutić J. Molecular detection and characterization of Porcine Kobuvirus in domestic pigs and wild boars in Serbia. Res Vet Sci. 2020;132:404–6.
24. Zhai SL, Zhang H, Lin T, Chen SN, Zhou X, Chen QL, et al. A novel porcine kobuvirus emerged in piglets with severe diarrhoea in China. Transbound Emerg Dis. 2017;64:1030–6.
25. Hu ZM, Yang Y Le, Xu LD, Wang B, Qin P, Huang YW. Porcine Torovirus (PToV)-A brief review of etiology, diagnostic assays and current epidemiology. Front Vet Sci. 2019;6:1–6.
26. Fujii Y, Kashima Y, Sunaga F, Aoki H, Imai R, Sano K, et al. Complete genome sequencing and genetic analysis of a Japanese porcine torovirus strain detected in swine feces. Arch Virol. Springer Vienna; 2020;165:471–7.
27. Shin DJ, Park SI, Jeong YJ, Hosmillo M, Kim HH, Kim HJ, et al. Detection and molecular characterization of porcine toroviruses in Korea. Arch Virol. 2010;155:417–22.
28. Pignatelli J, Jiménez M, Grau-Roma L, Rodríguez D. Detection of porcine torovirus by real time RT-PCR in piglets from a Spanish farm. J Virol Methods. 2010;163:398–404.
29. Zhou L, Wei H, Zhou Y, Xu Z, Zhu L, Horne J. Molecular epidemiology of Porcine torovirus (PToV) in Sichuan Province, China: 2011-2013. Virol J. 2014;11:1–9.
30. Qin P, Li H, Wang J, Wang B, Xie R, Xu H, et al. Genetic and pathogenic characterization of a novel reassortant mammalian orthoreovirus 3 (MRV3) from a diarrheic piglet and seroepidemiological survey of MRV3 in diarrheic pigs from east China Pan. Vet Microbiol. 2017;208:126–36.
31. Singh F, Rajukumar K, Senthilkumar D, Venkatesh G, Srivastava D, Kombiah S, et al. First report on co-isolation and whole-genomic characterisation of mammalian orthorubulavirus 5 and mammalian orthoreovirus type 3 from domestic pigs in India. Arch Virol. Springer Vienna; 2022;167:1529–45.
32. Day JM. The diversity of the orthoreoviruses: Molecular taxonomy and phylogentic divides. Infect Genet Evol. 2009;9:390–400.
33. Luo Y, Fei L, Yue H, Li S, Ma H, Tang C. Prevalence and genomic characteristics of a novel reassortment mammalian orthoreovirus type 2 in diarrhea piglets in Sichuan, China. Infect Genet Evol. Elsevier; 2020;85:104420.
34. Lelli D, Beato MS, Cavicchio L, Lavazza A, Chiapponi C, Leopardi S, et al. First identification of mammalian orthoreovirus type 3 in diarrheic pigs in Europe. Virol J. Virology Journal; 2016;13:1–5.
35. Kumthip K, Khamrin P, Kongkaew A, Vachirachewin R, Malasao R, Ushijima H, et al. Molecular epidemiology and characterization of porcine adenoviruses in pigs with diarrhea in Thailand. Infect Genet Evol. Elsevier; 2019;67:73–7.
36. De Motes CM, Clemente-Casares P, Hundesa A, Martín M, Girones R. Detection of Bovine and Porcine Adenoviruses for Tracing the Source of Fecal Contamination. Appl Environ Microbiol. 2004;70:1448–54.
37. Buitrago D, Cano-Gómez C, Agüero M, Fernandez-Pacheco P, Gómez-Tejedor C, Jiménez-Clavero MÁ. A survey of porcine picornaviruses and adenoviruses in fecal samples in Spain. J Vet Diagnostic Investig. 2010;22:763–6.
38. Jackova A, Sliz I, Mandelik R, Salamunova S, Novotny J, Kolesarova M, et al. Porcine kobuvirus 1 in healthy and diarrheic pigs: Genetic detection and characterization of virus and co-infection with rotavirus A. Infect Genet Evol. The Authors; 2017;49:73–7.
39. Kumthip K, Khamrin P, Saikruang W, Kongkaew A, Vachirachewin R, Ushijima H, et al. Detection and genetic characterization of porcine astroviruses in piglets with and without diarrhea in Thailand. Arch Virol. Springer Vienna; 2018;163:1823–9.
40. Zhou Y, Chen L, Zhu L, Xu Z. Molecular detection of porcine torovirus in piglets with diarrhea in southwest China. Sci World J. 2013;2013.
41. Kwon HJ, Kim HH, Kim HJ, Park JG, Son KY, Jung J, et al. Detection and molecular chracterization of porcine type 3 orthoreoviruses circulating in South Korea. Vet Microbiol. 2012;157:456–63.
42. Gunn L, Collins PJ, Fanning S, McKillen J, Morgan J, Staines A, et al. Detection and characterisation of novel bocavirus (genus Bocaparvovirus) and gastroenteritis viruses from asymptomatic pigs in Ireland. African J Disabil. 2015;5.
43. García-Hernández M-E, Trujillo-Ortega M-E, Alcaraz-Estrada S-L, Lozano-Aguirre- Beltrán L, Sandoval-Jaime C, Taboada-Ramírez BI, et al. Molecular Detection and Characterization of Porcine Epidemic Diarrhea Virus and Porcine Aichivirus C Coinfection in México. Viruses. 2021;13:738.
44. Nantel-Fortier N, Gauthier M, L’Homme Y, Lachapelle V, Fravalo P, Brassard J. The swine enteric virome in a commercial production system and its association with neonatal diarrhea. Vet Microbiol. Elsevier B.V.; 2022;266:109366.
45. Su M, Qi S, Yang D, Guo D, Yin B, Sun D. Coinfection and Genetic Characterization of Porcine Astrovirus in Diarrheic Piglets in China From 2015 to 2018. Front Vet Sci. 2020;7:3–6.
46. Salamunova S, Jackova A, Mandelik R, Novotny J, Vlasakova M, Vilcek S. Molecular detection of enteric viruses and the genetic characterization of porcine astroviruses and sapoviruses in domestic pigs from Slovakian farms. BMC Vet Res. BMC Veterinary Research; 2018;14:1–9.
47. Werid GM, Ibrahim YM, Chen H, Fu L, Wang Y. Molecular Detection and Genetic Characterization of Potential Zoonotic Swine Enteric Viruses in Northern China. Pathogens. 2022;11:28–30.
48. Chuchaona W, Khamrin P, Yodmeeklin A, Kongkaew A, Vachirachewin R, Kumthip K, et al. Detection and molecular characterization of porcine kobuvirus in piglets in 2009–2013 in northern Thailand. Trop Anim Health Prod. Tropical Animal Health and Production; 2017;49:1077–80.
49. Valkó A, Marosi A, Cságola A, Farkas R, Rónai Z, Dán Á. Frequency of diarrhoea-associated viruses in swine of various ages in Hungary. Acta Vet Hung. 2019;67:140–50.
50. Zhou W, Ullman K, Chowdry V, Reining M, Benyeda Z, Baule C, et al. Molecular investigations on the prevalence and viral load of enteric viruses in pigs from five European countries. Vet Microbiol. 2016;182:75–81.
51. Ulloa JC, Olarte-Aponte AM, Ospina JC, Rincon MA. Experimental infection of conventional newly-weaned piglets with porcine astrovirus. Acta Virol. 2019;63:96–102.
52. Fang Q, Wang C, Liu H, Wu Q, Liang S, Cen M, et al. Pathogenic characteristics of a porcine astrovirus strain isolated in China. Viruses. 2019;11:1–17.
53. Thimmasandra Narayanappa A, Sooryanarain H, Deventhiran J, Cao D, Ammayappan Venkatachalam B, Kambiranda D, et al. A novel pathogenic Mammalian orthoreovirus from diarrheic pigs and Swine blood meal in the United States. MBio. American Society for Microbiology (ASM); 2015;6:e00593-15.
54. Ducatelle R, Coussement W, Hoorens J. Sequential Pathological Study of Experimental Porcine Adenovirus Enteritis. Vet Pathol. 1982;19:179–89.
55. McAdaragh J, Eustis S, Benfield D. Adenovirus associated with diarrhea in pigs. Conf Res Work Anim Dis. Chicago; 1980.
56. Goecke NB, Hjulsager CK, Kongsted H, Boye M, Rasmussen S, Granberg F, et al. No evidence of enteric viral involvement in the new neonatal porcine diarrhoea syndrome in Danish pigs. BMC Vet Res. BioMed Central; 2017;13:315.
57. Alonso-Padilla J, Pignatelli J, Simon-Grifé M, Plazuelo S, Casal J, Rodríguez D. Seroprevalence of porcine torovirus (PToV) in Spanish farms. BMC Res Notes. 2012;5.
58. Carvajal A, Argüello H, Martínez-Lobo FJ, Costillas S, Miranda R, de Nova PJG, et al. Porcine epidemic diarrhoea: New insights into an old disease. Porc Heal Manag. Porcine Health Management; 2015;1:1–8.
59. Dortmans JCFM, Li W, van der Wolf PJ, Buter GJ, Franssen PJM, van Schaik G, et al. Porcine epidemic diarrhea virus (VDEP) introduction into a naive Dutch pig population in 2014. Vet Microbiol. 2018;221:13–8.
60. Kong F, Xu Y, Ran W, Yin B, Feng L, Sun D. Cold exposure-induced up-regulation of HSP70 positively regulates VDEP mRNA synthesis and protein expression in vitro. Pathogens. 2020;9.
61. Vidal A, Martín-valls GE, Tello M, Mateu E, Martín M. Prevalence of enteric pathogens in diarrheic and non diarrheic samples from pig farms with neonatal diarrhea in the North East of Spain. Vet Microbiol. 2019;237:108419.
62. Mesonero-Escuredo S, Strutzberg-Minder K, Casanovas C, Segalés J. Viral and bacterial investigations on the aetiology of recurrent pig neonatal diarrhoea cases in Spain. Porc Heal Manag. Porcine Health Management; 2018;4:1–6.
63. Cortey M, Díaz I, Vidal A, Martín-Valls G, Franzo G, Gómez De Nova PJ, et al. High levels of unreported intraspecific diversity among RNA viruses in faeces of neonatal piglets with diarrhoea. BMC Vet Res. BMC Veterinary Research; 2019;15:1–13.
64. Bauer E, Williams BA, Smidt H, Verstegen MWA, Mosenthin R. Influence of the gastrointestinal microbiota on development of the immune system in young animals. Curr Issues Intest Microbiol. 2006;7:35–52.
65. Qiu M, Li S, Xiao Y, Li J, Zhang Y, Li X, et al. Pathogenic and metagenomic evaluations reveal the correlations of porcine epidemic diarrhea virus, porcine kobuvirus and porcine astroviruses with neonatal piglet diarrhea. Microb Pathog. Elsevier Ltd; 2022;170:105703.
66. Anbalagan S, Peterson J, Wassman B, Elston J, Schwartz K. Genome sequence of torovirus identified from a pig with porcine epidemic diarrhea virus from the United States. Genome Announc. 2014;2:3–4.
67. Dufkova L, Scigalkova I, Moutelikova R, Malenovska H, Prodelalova J. Genetic diversity of porcine sapoviruses, kobuviruses, and astroviruses in asymptomatic pigs: An emerging new sapovirus GIII genotype. Arch Virol. 2013;158:549–58.
68. Mor SK, Chander Y, Marthaler D, Patnayak DP, Goyal SM. Detection and molecular characterization of Porcine astrovirus strains associated with swine diarrhea. J Vet Diagnostic Investig. 2012;24:1064–7.
69. Cai Y, Yin W, Zhou Y, Li B, Ai L, Pan M, et al. Molecular detection of Porcine astrovirus in Sichuan Province, China. Virol J. 2016;13:1–5.
70. Lee MH, Jeoung HY, Park HR, Lim JA, Song JY, An DJ. Phylogenetic analysis of porcine astrovirus in domestic pigs and wild boars in South Korea. Virus Genes. 2013;46:175–81.
71. Tamura K, Stecher G, Kumar S. MEGA11: Molecular Evolutionary Genetics Analysis Version 11. Mol Biol Evol. 2021;38:3022–7.
72. Liu Q, Wang HY. Porcine enteric coronaviruses: an updated overview of the pathogenesis, prevalence, and diagnosis. Vet Res Commun. Springer Netherlands; 2021;45:75–86.
73. Saif LJ, Wang Q, Vlasova AN, Jung K, Xiao S. Coronaviruses. In: Zimmerman JJ, Karriker LA, Ramirez A, Schwartz KJ, Stevenson GW, Zhang J, editors. Dis swine. 11th ed. Hoboken: John Wiley and Sons; 2019. p. 488–523.
74. Li C, Lu H, Geng C, Yang K, Liu W, Liu Z, et al. Epidemic and Evolutionary Characteristics of Swine Enteric Viruses in South Central China from 2018 to 2021. Viruses. 2022;14.
75. Wang L, Byrum B, Zhang Y. Detection and genetic characterization of deltacoronavirus in pigs, Ohio, USA, 2014. Emerg Infect Dis. Centers for Disease Control and Prevention; 2014;20:1227–30.
76. Kong F, Wang Q, Kenney SP, Jung K, Vlasova AN, Saif LJ. Porcine Deltacoronaviruses: Origin, Evolution, Cross Species Transmission and Zoonotic Potential. Pathogens. 2022;11:1–17.
77. Xu Z, Zhang Y, Gong L, Huang L, Lin Y, Qin J, et al. Isolation and characterization of a highly pathogenic strain of Porcine enteric alphacoronavirus causing watery diarrhoea and high mortality in newborn piglets. Transbound Emerg Dis. 2018;66:119–30.
78. Fu X, Fang B, Liu Y, Cai M, Jun J, Ma J, et al. Newly emerged porcine enteric alphacoronavirus in southern China: Identification, origin and evolutionary history analysis. Infect Genet Evol. Elsevier; 2018;62:179–87.
79. Zhou P, Fan H, Lan T, Yang X Lou, Shi WF, Zhang W, et al. Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin. Nature. 2018;556:255–9.
80. Marthaler D, Homwong N, Rossow K, Culhane M, Goyal S, Collins J, et al. Rapid detection and high occurrence of porcine rotavirus A, B, and C by RT-qPCR in diagnostic samples. J Virol Methods. Elsevier B.V.; 2014;209:30–4.
81. Wu FT, Liu LTC, Jiang B, Kuo TY, Wu CY, Liao MH. Prevalence and diversity of rotavirus A in pigs: Evidence for a possible reservoir in human infection. Infect Genet Evol. Elsevier B.V.; 2022;98:105198.
82. Chepngeno J, Takanashi S, Diaz A, Michael H, Paim FC, Rahe MC, et al. Comparative Sequence Analysis of Historic and Current Porcine Rotavirus C Strains and Their Pathogenesis in 3-Day-Old and 3-Week-Old Piglets. Front Microbiol. 2020;11:1–13.
83. Qin P, Li H, Wang JW, Wang B, Xie RH, Xu H, et al. Genetic and pathogenic characterization of a novel reassortant mammalian orthoreovirus 3 (MRV3) from a diarrheic piglet and seroepidemiological survey of MRV3 in diarrheic pigs from east China. Vet Microbiol. 2017;208:126–36.
[/registrados]
