
Para leer más contenidos de porciSapiens Julio 2021
Para leer más contenidos de porciSapiens Julio 2021
 Un aspecto intrigante en la identificación de estas moléculas como biomarcadores se deriva de su papel en la comunicación entre células, su secreción activa desde las células al medio extracelular, su alta estabilidad en los fluidos corporales y su facilidad de obtención. Todas estas características confieren a los miARNs el potencial para convertirse en una herramienta no invasiva que permita evaluar el bienestar animal en condiciones de estrés metabólico, ambiental y de manejo. Esta revisión ofrece una visión general de los conocimientos actuales sobre el uso potencial de miARNs tisulares y/o circulantes como biomarcadores para la evaluación del estado de salud y bienestar en el porcino. |
NUEVOS PARÁMETROS DE EVALUACIÓN DEL BIENESTAR ANIMAL

Recientemente, el protocolo Welfare Quality introdujo el uso de parámetros basados en los animales centrados en sus necesidades e incluyendo la evaluación de indicadores adecuados (válidos, fiables y viables) que permitan evaluar su bienestar físico y mental1.
El deterioro del bienestar de los animales suele estar causado por el estrés crónico resultante de la incapacidad para hacer frente a los factores ambientales combinado con la vulnerabilidad genética (por ejemplo, la concentración de neurotransmisores como la serotonina y la respuesta inmunitaria individual)2-4.
Según la bibliografía actual, los indicadores de bienestar del ganado se clasifican en tres categorías principales5-8:
![]() Parámetros fisiológicos
Parámetros fisiológicos
![]() Observaciones del comportamiento
Observaciones del comportamiento
![]() Calidad del producto
Calidad del producto
Los parámetros fisiológicos, incluidos los sanguíneos9-11 y la observación del comportamiento permiten evaluar el bienestar animal in vivo12.
La perturbación metabólica sistémica resultante del estrés crónico también se ha investigado mediante la metabolómica y ha permitido la identificación de parámetros directamente asociados a las condiciones de manejo y alojamiento, y regulados por el eje hipotálamo-hipófisissuprarrenal (HHA)13,14. Otros estudios han tratado de identificar hormonas y otras moléculas encontradas en niveles fuera de los “rangos fisiológicos”15-17.
MIARNS COMO POTENCIALES BIOMARCADORES
Los miARN son[registrados] segmentos cortos de ARN (aproximadamente 22 nucleótidos) que no codifican para proteínas. Codificados en el genoma de la célula, son trascritos en el núcleo, dando lugar a los pri-miARN que son procesados y exportados al citoplasma para realizar su función como miARN maduros18.
La función regulatoria de la expresión génica postranscripcional de los miARN reside en su capacidad para unirse a la región 3’UTR del ARN mensajero (ARNm) del gen diana.
De esta forma, la unión de un miARN a un segmento complementario de ARNm se traduce en una disminución de la síntesis de la proteína codificada por el gen diana18.
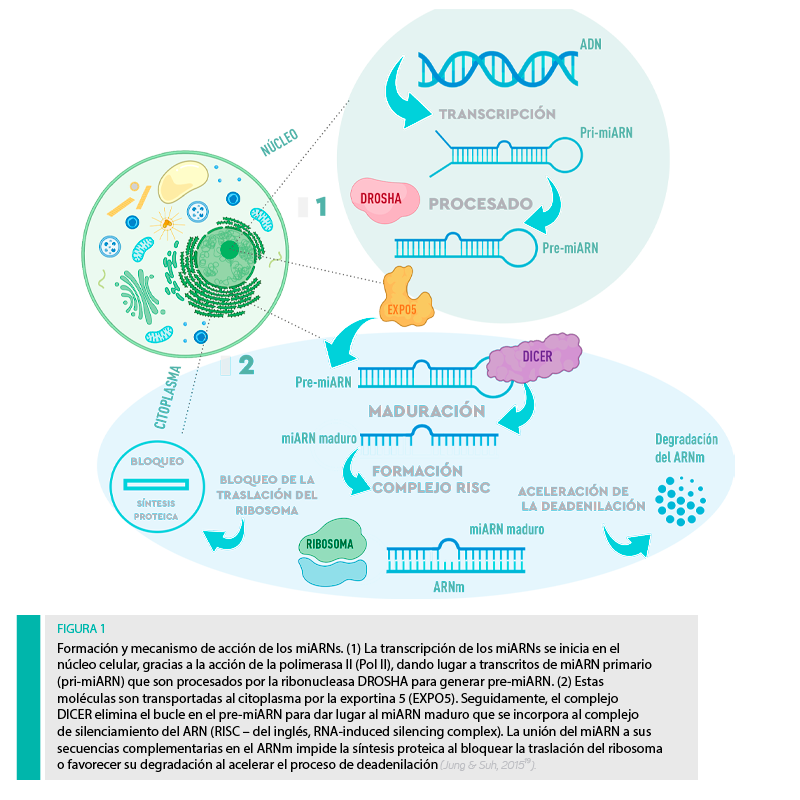
Los miARNs coordinan procesos celulares, incluyendo la modulación del desarrollo del animal, la homeostasis, las respuestas inmunitarias y el control de las infecciones, y también son cruciales para la regulación de la renovación de las células madre y la diferenciación de los tejidos20-23.
Tras un estímulo fisiológico o una lesión, los miARNs circulantes (c-miARNs) pueden ser liberados por las células a la sangre o a otros fluidos corporales de forma activa (secreción) o pasiva (filtración a través de la membrana)24-26.
Por ello, parte del interés suscitado por los c-miARNs se relaciona con su implicación en la regulación de vías moleculares en las células receptoras y su notable potencial como biomarcadores de enfermedades y trastornos27.
Algunos de los aspectos más interesantes de la identificación de moléculas de miARN como biomarcadores se derivan de su28:
![]() Expresión espacial y temporal altamente regulada
Expresión espacial y temporal altamente regulada
![]() Secreción activa desde las células al entorno extracelular
Secreción activa desde las células al entorno extracelular
![]() Gran estabilidad en los fluidos corporales
Gran estabilidad en los fluidos corporales
BIOMARCADORES DE ESTADOS FISIOLÓGICOS
![]()
Los perfiles de los c-miARNs cambian en el caso de enfermedades e infecciones víricas o bacterianas29-31, y según el estado fisiológico (por ejemplo, la gestación)32,33, lo que implica que estas moléculas son biomarcadores adecuados para monitorizar diferentes condiciones fisiológicas en los animales.
BIOMARCADORES DE ESTRÉS
![]()
Evidencias obtenidas en modelos de roedores indican que los c-miARNs también pueden servir como biomarcadores de resiliencia o vulnerabilidad al estrés34, ya que los miARNs contribuyen a numerosos aspectos de la neurogénesis, la plasticidad neuronal y la respuesta al estrés, a la vez que modulan la expresión de genes implicados en el estrés psicosocial crónico35,36.
Relacionar las condiciones estresantes y sus efectos psicológicos es particularmente difícil en los animales de granja, pero se ha demostrado la asociación entre el estrés crónico y los conflictos sociales, el aislamiento y el hacinamiento37-39.
Hasta la fecha, ningún estudio ha investigado el papel de los miARN en los mecanismos moleculares que subyacen a las respuestas al estrés psicológico en las especies ganaderas, pero, dado el inherente carácter social de estos animales, la capacidad de estas moléculas para modular las redes moleculares asociadas al estrés mental merece una mayor atención.
DETECCIÓN DE MIARN – DEL LABORATORIO AL CAMPO
![]()
Debido al alto nivel de homología de las secuencias, los miARNs pueden ser detectados fácilmente sin necesidad de utilizar anticuerpos o ensayos específicos para cada especie. Por ello, la identificación de miARNs adecuados es un interesante campo de investigación que puede proporcionar una visión más amplia del bienestar y la salud de los animales a nivel molecular.
Los c-miARNs se encuentran entre los biomarcadores clínicos más prometedores para la identificación de trastornos relacionados con el estrés en los animales, siendo herramientas válidas para evaluar el bienestar de un animal a lo largo de su vida, además de permitir puntuar la calidad de sus productos a lo largo de la cadena de suministro de alimentos.
Recientemente, se ha propuesto restringir el concepto de estrés a “las condiciones en las que una demanda ambiental excede la capacidad natural de regulación de un organismo, en situaciones particulares que incluyen lo impredecible e incontrolable”, y debe estar estrictamente relacionado con una condición de salud40. Dentro de estas situaciones “imprevisibles e incontrolables” pueden incluirse varios eventos desafiantes durante la vida productiva de los animales:
![]() Estrés inmunológico y metabólico
Estrés inmunológico y metabólico
![]() Estrés asociado al manejo y la manipulación
Estrés asociado al manejo y la manipulación
![]() Estrés por separación y reagrupación de los animales durante su ciclo de producción
Estrés por separación y reagrupación de los animales durante su ciclo de producción
![]() Estrés asociado al destete
Estrés asociado al destete
![]() Estrés asociado a cambios en la dieta
Estrés asociado a cambios en la dieta
![]() Estrés asociado al transporte
Estrés asociado al transporte
![]() Estrés ambiental
Estrés ambiental
MIARNS RELACIONADOS CON LA SALUD Y EL ESTRÉS EN CERDOS
Muchos de los efectos de los factores estresantes en las granjas porcinas son evidenciables, por ejemplo, la expresión irregular del celo, el aumento de las tasas de aborto, la reducción de la vitalidad de los lechones o el aumento de infecciones. Los efectos del estrés a lo largo del ciclo vital de los cerdos implican cambios fisiológicos que afectan a su salud y bienestar (Figura 2).
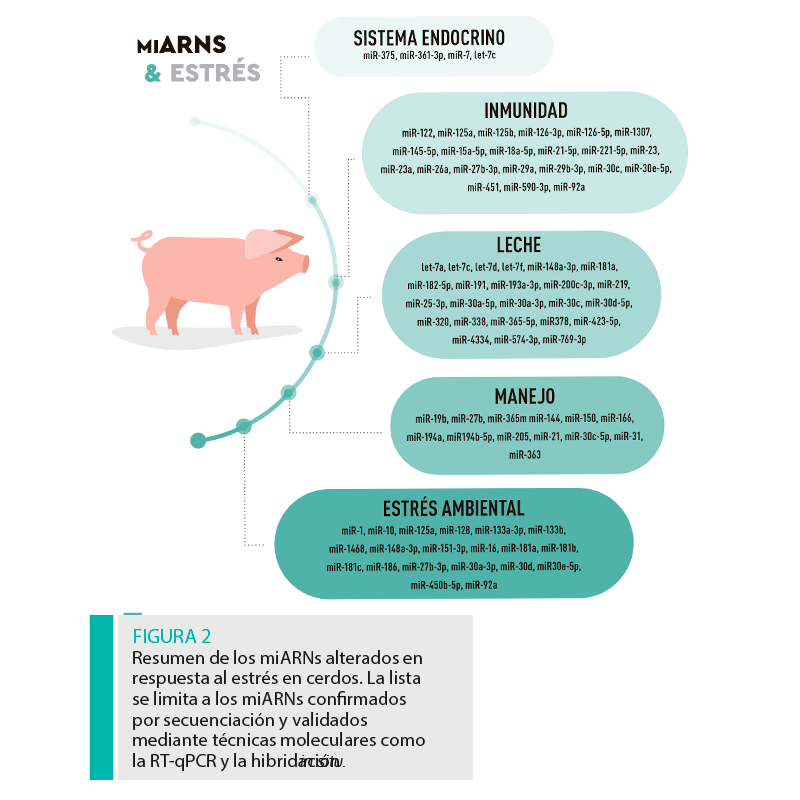
EL SISTEMA ENDOCRINO Y LA RESPUESTA AL ESTRÉS
![]()
La primera reacción frente a un factor estresante es una alteración adaptativa del eje HHA. Sin embargo, el estrés persistente puede inducir cambios en las redes de regulación génica, incluyendo alteraciones en los niveles de los miARNs41.

MIARN VS CRH
Ante un evento estresante, la hormona liberadora de corticotropina (CRH) activa al eje HHA, promoviendo la secreción de la hormona adrenocorticotropa (ACTH) por parte de la hipófisis y de glucocorticoides por parte de las glándulas adrenales.
En este sentido, la relación entre la CRH y miR-375 en la regulación de la síntesis de catecolaminas se ha estudiado en la glándula adrenal de cerdas.
Los resultados demuestran que la CRH inhibe la expresión de miR-375, que a su vez inhibe la expresión de catecolaminas42.
Además, el miR-375 tiene como diana al gen SP1, un efector descendente de la vía de la proteína quinasa A, disminuyendo la esteroidogénesis y la producción de glucocorticoides42.
MIARN VS FSH
Ye et al. investigaron la capacidad de los miARNs para regular post-transcripcionalmente la expresión in vitro de la hormona foliculoestimulante (FSH) en células de la hipófisis anterior de cerdas43, demostrando que los niveles de expresión de miR-361-3p y la abundancia de FSHB están correlacionados negativamente.
Tras la estimulación de las células con la hormona liberadora de gonadotropina (GnRH), el nivel de miR-361-3p disminuyó, mientras que el de FSHB aumentó. El mecanismo subyacente está relacionado con la capacidad de miR-361-3p para unirse directamente a la UTR 3’ del gen FSHB43.
MIARN VS ZEN
La capacidad de los miARNs para modular la expresión de la FSH también se ha reportado en el contexto de la exposición a zearalenona (ZEN), una micotoxina no esteroidea producida por Fusarium que se puede encontrar en los piensos y que provoca alteraciones en el sistema reproductor porcino44.
Los efectos de la ZEN en la hipófisis incluyeron la modulación de miR-7, que actúa como regulador de la síntesis y secreción de gonadotropinas. Los autores demostraron que la ZEN aumentó la expresión de miR-7, inhibiéndose la expresión de FSH al actuar directamente sobre el gen FOS44.
MIARN VS GH
Qi et al. exploraron los cambios en la expresión de miARN tras la estimulación de células hipofisarias porcinas con hormona liberadora de la hormona del crecimiento (GHRH) y con cortistatina e identificaron 19 y 35 miARNs con expresión diferencial, respectivamente.
Las pruebas funcionales demostraron que let-7c modulaba la expresión de los genes de la hormona del crecimiento 1 (GH1) y del receptor de la hormona liberadora de la hormona del crecimiento (GHRHR) uniéndose a sus respectivos 3’UTRs y promoviendo una disminución de la secreción de GH45.
INMUNIDAD
![]()
Varios estudios han analizado la implicación de los miARNs en la respuesta inmunitaria de los cerdos frente a virus, bacterias y parásitos, mientras que otros han comparado cerdos sanos de diferentes razas utilizando métodos de secuenciación, microarrays y RT-qPCR.
Los genes a los que se dirigen los miARNs identificados en estos estudios mediante predicción in silico o mediante validación experimental están implicados en vías relacionadas con la inmunidad, como la apoptosis, la inhibición de la replicación viral y el reconocimiento viral46-51.
En este apartado, nos centramos en los estudios que incluyeron la validación experimental de los genes diana y demostraron el potencial de los miARN como biomarcadores o para su uso en la terapia antiviral.
MIARN VS INFLUENZA
El papel de los miARNs como reguladores de la respuesta inmunitaria innata inducida por el virus de la Influenza A H1N2 (IAV) en los pulmones y los leucocitos se ha investigado en un modelo porcino in vivo46,47,52.
Brogaard et al. demostraron que varios miARNs y genes relacionados con el sistema inmunitario presentan una expresión diferencial en los pulmones y los leucocitos de los cerdos infectados46,47. En los pulmones, los autores correlacionaron la expresión de algunos miARNs y genes47:
![]() miR-29b-3p y miR-15a-5pdesempeñan un papel en la apoptosis al afectar la expresión de los genes BCL2 y MCL1.
miR-29b-3p y miR-15a-5pdesempeñan un papel en la apoptosis al afectar la expresión de los genes BCL2 y MCL1.
![]() miR21-5pmodula la expresión de citoquinas pro- y antiinflamatorias.
miR21-5pmodula la expresión de citoquinas pro- y antiinflamatorias.
![]() miR-18a-5p y miR- 590-3p actúan sobre el gen del factor de iniciación de la traducción eucariótica 2 alfa quinasa 2 (EIF2AK2) que codifica un inhibidor de la replicación viral.
miR-18a-5p y miR- 590-3p actúan sobre el gen del factor de iniciación de la traducción eucariótica 2 alfa quinasa 2 (EIF2AK2) que codifica un inhibidor de la replicación viral.
En los leucocitos, se detectaron diferentes miARNs en tres momentos tras la infección y el análisis de las interacciones entre miARNs y genes demostró que estaban implicados en la apoptosis y en las respuestas inmunitarias innatas47.
miR-29B-3P Y miR-21-5P REPRESENTAN POSIBLES DIANAS CANDIDATAS PARA LA MODULACIÓN DE LA RESPUESTA INMUNITARIA FRENTE AL VIRUS INFLUENZA A H1N1
MIARN VS PRRS
Se ha investigado ampliamente la capacidad de los miARNs para modular la respuesta inmunitaria durante la infección por el PRRSV.
![]() miR-27b-3p y miR-26ainhiben la replicación del PRRSV al actuar sobre la proteína no estructural 2 del virus en los macrófagos alveolares50, afectando también a factores implicados en la vía del IFN, incluidos los genes MX1 e IFI44/IFI44, las quimioquinas, las citoquinas y el factor del complemento en las células MARC-14553.
miR-27b-3p y miR-26ainhiben la replicación del PRRSV al actuar sobre la proteína no estructural 2 del virus en los macrófagos alveolares50, afectando también a factores implicados en la vía del IFN, incluidos los genes MX1 e IFI44/IFI44, las quimioquinas, las citoquinas y el factor del complemento en las células MARC-14553.
![]() miR-29aregula la replicación del PRRSV al unirse directamente a su ARN genómico y promover su replicación mediante su unión al extremo 3’UTR del gen AKT348.
miR-29aregula la replicación del PRRSV al unirse directamente a su ARN genómico y promover su replicación mediante su unión al extremo 3’UTR del gen AKT348.
![]() miR-23 aumenta la expresión de Interferón tipo I (IFN-1) al actuar sobre los genes IRF3/IRF7, lo que puede contribuir a la supresión de la infección por el PRRSV54.
miR-23 aumenta la expresión de Interferón tipo I (IFN-1) al actuar sobre los genes IRF3/IRF7, lo que puede contribuir a la supresión de la infección por el PRRSV54.
![]() miR-30cpuede ser estimulado por el PRRSV para impedir la señalización mediada por IFN-I al unirse directamente a los genes JAK1 y de la cadena β del receptor de interferón α/β, promoviendo así la replicación viral55.
miR-30cpuede ser estimulado por el PRRSV para impedir la señalización mediada por IFN-I al unirse directamente a los genes JAK1 y de la cadena β del receptor de interferón α/β, promoviendo así la replicación viral55.
MIARN VS PESTE PORCINA AFRICANA
El efecto de la infección por el virus de la peste porcina africana (VPPA) sobre la expresión diferencial de miARNs de los cerdos se ha investigado in vivo comparando su expresión en animales infectados con dos cepas virales (virulenta vs atenuada)56.
Se seleccionaron diez miARNs para la predicción de dianas basándose en la mayor representación en tejidos: miR-23a, miR-30e-5p, miR-92a, miR-122, miR-125b, miR-126-5p, miR-145-5p, miR-125a, miR-451 y miR-126-3p.
Los potenciales genes diana estaban relacionados con la respuesta inmunitaria, afectando a las vías de señalización de los receptores de linfocitos B y T, la citotoxicidad mediada por células NK o la fagocitosis mediada por receptores Fc gamma, así como a procesos relacionados con la patogénesis de la infección y la interacción virus-huésped56.
MIARN VS FIEBRE AFTOSA
Se ha descrito que la expresión del miR-1307 se encuentra aumentada en células renales porcinas en relación a una preactivación y potenciación de la señalización del sistema inmunitario innato en la fase inicial de la infección por el virus de la Fiebre Aftosa (VFA). Este miARN promueve la degradación de la proteína viral estructural VP3 a través de la vía del proteasoma y la regulación de genes relacionados con el sistema inmunitario51.
El potencial terapéutico del miR-1307 también se ha demostrado mediante la inyección subcutánea de miR-1307 agomir en ratones lactantes, donde retrasó la letalidad inducida por el VFA51.
MIARN VS DIARREA EPIDÉMICA PORCINA
El aumento de la expresión del miR-221-5p en células MARC-145 infectadas con el virus de la Diarrea Epidémica Porcina (PEDV) se relaciona con la inhibición de la replicación viral al actuar directamente sobre su 3’UTR, además de mejorar la respuesta inmunitaria del hospedador49.
LECHE
![]()
Los exosomas presentes en la leche pueden transportar miARN desde las células donantes a las receptoras, regulando así la expresión de los genes diana y la función de las receptoras, pudiendo llegar a diferentes tejidos tras su absorción intestinal57.
MIARN – CALOSTRO VS LECHE
Gu et al. investigaron el perfil de expresión de miARNs en cerdas a lo largo de toda la fase de lactación (desde el parto hasta los 28 días), centrándose en la expresión de miARNs relacionados con el sistema inmunitario58.
Demostraron que los miARNs relacionados con el sistema inmunitario (miR-148a-3p, miR- 182-5p, miR-200c-3p, miR-25-3p, miR-30a-5p, miR-30d-5p y miR-574-3p) estaban presentes en mayor cantidad en el calostro que en la leche, y también eran más abundantes en la sangre de los lechones alimentados sólo con calostro en comparación con los alimentados sólo con leche58.
Los miARNs liberados por los exosomas de la leche porcina también han sido caracterizados mediante secuenciación y los 10 miARNs más abundantes identificados han sido: miR-193a-3p, miR- 423-5p, miR-320, miR-181a, miR-30a-3p, miR-378, miR-191, let-7a, let-7f, y let-7c59.

MIARN VS POLISACÁRIDOS DE GINSENG
Se ha investigado el efecto de la suplementación dietética con polisacáridos de ginseng (GPS) sobre los miARNs de la leche para evaluar su capacidad para modular la respuesta inmunitaria de los lechones.
Se identificaron 10 miARNs que tenían una expresión aumentada y 16 que tenían expresión reducida en el grupo tratado con GPS, sugiriéndose que estos miARNs actúan como potenciales reguladores de las funciones inmunitarias60.
En este caso, los niveles de expresión de miR-30a y let-7d también se vieron influidos por la suplementación con GPS, lo que vuelve a poner de manifiesto su papel esencial en la leche.
MIARN VS LIPOPOLISACÁRIDO
Xie et al. demostraron el efecto protector de los miARNs de la leche porcina investigando el papel de miR-181a, miR-30c, miR- 365-5p y miR-769-3p en los exosomas de la leche. La elevada expresión de estos miARNs se correlacionó con una disminución significativa de los niveles de expresión de sus genes diana y de las proteínas codificadas que participan en la vía de p5361.
Estos autores habían demostrado previamente que, tras la exposición al lipopolisacárido (LPS), miR-4334, miR-219 y miR-338 actuaban sobre los genes TLR4, MyD88 y TP53, respectivamente, reduciendo así la apoptosis inducida por el LPS a través de las vías TLR4/NF-kB y p5361,62.
MANEJO
![]()
MIARN VS CASTRACIÓN & CORTE DE COLAS
Los procedimientos rutinarios de manejo de los lechones, como la castración y el corte de colas, son eventos asociados al dolor agudo que amenazan su bienestar.
En un estudio reciente y por primera vez en una especie porcina, se estudiaron las concentraciones de miARNs en saliva y se evaluaron los niveles de expresión de miR-19b, miR-27b-3p, miR-215, miR-22-3p, miR-155-5p, miR-365-5p y miR- 204 para determinar su potencial como biomarcadores del dolor mediante RT-qPCR26, demostrando que estos miARNs eran más abundantes en los animales en los que el dolor no se mitigaba mediante un anestésico, por lo que podrían ser potenciales biomarcadores para la identificación del dolor en estos casos.
miR-19B, miR-365 Y miR-27B SE CORRELACIONARON CON LA INFLAMACIÓN RESULTANTE DE LA CASTRACIÓN Y EL CORTE DE COLAS SIN ANESTESIA26
MIARN VS DESTETE
Para dilucidar el papel de los miARNs en relación al estrés del destete, Tao y Xu63 compararon los perfiles de miARN del yeyuno y del suero procedentes de lechones durante la lactación y tras el destete.
Se identificó un gran número de miARNs con expresión diferencial en los días 1, 4 y 7, muchos de ellos involucrados en la modulación del metabolismo del intestino delgado, las respuestas al estrés y las funciones inmunitarias63.
Tres c-miARNs (miR-21, miR-31 y miR-205) mostraron una expresión aumentada y 7 (miR-30c-5p, miR-144, miR-150, miR-186, miR-194a, miR-194b-5p y miR-363) presentaron una expresión disminuida en lechones destetados en comparación con los lechones lactantes.
Dado que la expresión de miR-194b-5p se redujo significativamente en el suero y en el intestino delgado de los lechones destetados, los autores plantearon la hipótesis de que su expresión en el suero podría reflejar la disminución de la expresión en el intestino delgado.
Este hallazgo es importante dado que se ha demostrado que el miR-194b-5p inhibe la expresión del pequeño modificador similar a la ubiquitina 2 (SUMO2)64. SUMO2 es fundamental para la homeostasis celular en situaciones de estrés endógeno o ambiental, incluyendo, entre otros, el choque térmico y el estrés nutricional65.
miR-194B-5P PODRÍA SER UN CANDIDATO A BIOMARCADOR PARA CONTROLAR LA SALUD INTESTINAL DE LOS LECHONES DURANTE EL PERIODO DE DESTETE
ESTRÉS AMBIENTAL
![]()
MIARN VS ESTRÉS TÉRMICO
Al carecer los cerdos de glándulas sudoríparas y tener unos pulmones relativamente pequeños en comparación con su masa corporal, son más sensibles al calor que otras especies ganaderas66. Además, las nuevas líneas genéticas de cerdos producen casi un 20% más de calor que las razas anteriores67.
El estrés térmico afecta a varios aspectos de la producción porcina, como el rendimiento reproductivo, el consumo de pienso, la condición corporal, la respuesta inmunitaria y la producción de leche68-72, y afecta al desarrollo muscular, modificando el equilibrio entre la síntesis y la degradación de proteínas73.
Hao et al. investigaron cómo el estrés térmico afectaba al perfil de expresión de miARNs musculares de los cerdos74. Los cerdos fueron criados a una temperatura ambiental constante moderada (22˚C) o alta (30˚C) durante 21 días. La secuenciación de las muestras recogidas del músculo longísimo puso de relevancia a 58 miARNs con expresión diferencial, 30 con una expresión reducida y 28 con una expresión aumentada74.
De los genes a los que potencialmente se dirigían los miARNs con expresión diferencial, se analizaron aquellos relacionados con el metabolismo muscular y la respuesta al estrés, y se descubrió que la expresión de la piruvato deshidrogenasa quinasa 4 (PDK4), la proteína de choque térmico 90 (Hsp90), la desmina (DES), la lactato deshidrogenasa A (LDHA) y la estearoil-CoA desaturasa (SCD) presentaban una correlación inversa con los miARNs con expresión diferencial74.
Estos resultados demuestran que el estrés térmico puede modular la expresión de los miARNs en el músculo y la glándula mamaria, influyendo en la estructura y función de los tejidos, la glucólisis y el metabolismo del lactato y los lípidos.
El bienestar de los animales y su rendimiento productivo pueden verse afectados, lo que implica que la identificación y validación de los miARN y sus genes diana es esencial para el desarrollo de nuevas estrategias de reproducción basadas en genes reguladores.

CONCLUSIONES
Los estudios realizados hasta la fecha se han centrado en gran medida en los biomarcadores de estrés o resiliencia, y muchos de ellos se han centrado en los miARNs que pueden proporcionar información sobre la regulación de diferentes procesos.
Los biomarcadores convencionales pueden ser útiles como marcadores de estrés y lesiones, pero proporcionan información limitada sobre el mecanismo celular que subyace a la adaptación de los animales a los acontecimientos adversos.
Los miARNs extracelulares pueden combinarse con otras mediciones fenotípicas para monitorizar con mayor precisión las respuestas al estrés de animales, permitiendo a los productores monitorizar los cambios en el manejo de los animales o en los sistemas de producción, determinando si estos cambios pueden reducir o eliminar los efectos fisiológicos del estrés.
La falta de un protocolo estándar para la cuantificación de los c-miARNs limita la comparación de los perfiles de expresión de miARNs entre diferentes laboratorios, por lo que el reconocimiento de métodos estandarizados comunes para minimizar los posibles sesgos sigue siendo una cuestión crítica.
Para implementar los c-miARNs como nuevos biomarcadores, debe establecerse su origen celular y su relación con sus tejidos, así como su relación con los factores de estrés.
En un segundo paso, será necesario realizar estudios a gran escala para comparar la expresión de los miARN en las especies ganaderas en condiciones de estrés de duración y fuerza variables, a fin de validar la aplicabilidad de los miARN como biomarcadores.
| En cerdos ya se han identificado varios miARNs con potencial como biomarcadores para la gestión y regulación del estrés y el control de la salud animal. A pesar de que la investigación sobre los miARNs en las especies ganaderas va por detrás de la de los humanos, estas moléculas tienen un gran potencial como nueva clase de biomarcadores relacionados con el estrés y la salud y representan un nuevo y apasionante campo para evaluar la gestión y el bienestar de los animales. |
Artículo traducido y adaptado de: Miretti, S., Lecchi, C., Ceciliani, F., & Baratta, M. (2020). MicroRNAs as Biomarkers for Animal Health and Welfare in Livestock. Frontiers In Veterinary Science, 7. doi: 10.3389/ fvets.2020.578193. (CC BY 4.0)
BIBLIOGRAFÍA
1. Welfare Quality R Consortium, Lelystad N. Welfare Quality Protocols. (2009). Available online at: http://www.welfarequalitynetwork.net/en-us/reports/assessment-protocols/ (accessed June 15, 2020).
2. Scharf SH, Schmidt MV. Animal models of stress vulnerability and resilience in translational research. Curr Psychiatry Rep. (2012) 14:159–65. doi: 10.1007/s11920-012-0256-0
3. Ellen ED, Bas Rodenburg T, Albers GAA, Elizabeth Bolhuis J, Camerlink I, Duijvesteijn N, et al. The prospects of selection for social genetic effects to improve welfare and productivity in livestock. Front Genet. (2014) 5:377. doi: 10.3389/fgene.2014.00377
4. Colditz IG, Hine BC. Resilience in farm animals: biology, management, breeding and implications for animal welfare. Anim Prod Sci. (2016) 56:1961–83. doi:10.1071/AN15297
5. Sánchez-Hidalgo M, Bravo V, Gallo C. Behavior and health indicators to assess cull cow’s welfare in livestock markets. Front Vet Sci. (2020) 7:471. doi:10.3389/fvets.2020.00471
6. Munoz CA, Campbell AJD, Hemsworth PH, Doyle RE. Evaluating the welfare of extensively managed sheep. PLoS ONE. (2019) 14:e0218603. doi: 10.1371/journal. pone.0218603
7. Vasdal G, Granquist EG, Skjerve E, De Jong IC, Berg C, Michel V, et al. Associations between carcass weight uniformity and production measures on farm and at slaughter in commercial broiler flocks. Poult Sci. (2019) 98:4261–8. doi: 10.3382/ps/pez252
8. Chulayo AY, Muchenje V. Activities of some stress enzymes as indicators of slaughter cattle welfare and their relationship with physico-chemical characteristics of beef. Animal. (2017) 11:1645–52. doi: 10.1017/S1751731117000222
9. Chen Y, Stookey J, Arsenault R, Scruten E, Griebel P, Napper S. Investigation of the physiological, behavioral, and biochemical responses of cattle to restraint stress. J Anim Sci. (2016) 94:3240–54. doi: 10.2527/jas.2016-0549
10. Wein Y, Geva Z, Bar-Shira E, Friedman A. Transport- related stress and its resolution in Turkey pullets: activation of a pro-inflammatory response in peripheral blood leukocytes. Poult Sci. (2017) 96:2601–13. doi:
10.3382/ps/pex076
11. Sejian V, Bhatta R, Gaughan JB, Dunshea FR, Lacetera N. Review: adaptation of animals to heat stress. Animal. (2018) 12:S431–44. doi: 10.1017/S1751731118001945
12. Grandin T, Shivley C. How farm animals react and perceive stressful situations such as handling, restraint, and transport. Animals. (2015) 5:1233–51. doi:10.3390/ani5040409
13. Qu H, Ajuwon KM. Metabolomics of heat stress response in pig adipose tissue reveals alteration of phospholipid and fatty acid composition during heat stress. J Anim Sci. (2018) 96:3184–95. doi: 10.1093/jas/sky127
14. Li J, Wijffels G, Yu Y, Nielsen LK, Niemeyer DO, Fisher AD, et al. Altered fatty acid metabolism in long duration road transport: an NMR-based metabonomics study in sheep. J Proteome Res. (2011) 10:1073–87. doi: 10.1021/pr100862t
15. Cornale P, Macchi E, Miretti S, Renna M, Lussiana C, Perona G, et al. Effects of stocking density and environmental enrichment on behavior and fecal corticosteroid levels of pigs under commercial farm conditions. J Vet Behav Clin Appl Res. (2015) 10:569–76. doi:10.1016/j.jveb.2015.05.002
16. Fustini M, Galeati G, Gabai G, Mammi LE, Bucci D, Baratta M, et al. Overstocking dairy cows during the dry period affects dehydroepiandrosterone and cortisol secretion. J Dairy Sci. (2017) 100:620–8. doi: 10.3168/jds.2016-11293
17. Marco-Ramell A, Arroyo L, Peña R, Pato R, Saco Y, Fraile L, et al. Biochemical and proteomic analyses of the physiological response induced by individual housing in gilts provide new potential stress markers. BMC Vet Res. (2016) 12:265. doi: 10.1186/s12917-016-0887-1
18. Díaz Carrasco, I., Guisado Rasco, A., & Ordoñez Fernández, A. (2016). ¿Qué son los micro RNA? ¿Para qué sirven? ¿Qué potenciales beneficios podrían tener en el contexto asistencial?. Cardiocore, 51(4), 161-166. doi: 10.1016/j.carcor.2015.02.002
19. Jung, H., & Suh, Y. (2015). Regulation of IGF -1 signaling by microRNAs. Frontiers In Genetics, 5. doi: 10.3389/fgene.2014.00472
20. Zhang Z, Cao Y, Zhai Y, Ma X, An X, Zhang S, et al. MicroRNA-29b regulates DNA methylation by targeting Dnmt3a/3b and Tet1/2/3 in porcine early embryo development. Dev Growth Differ. (2018) 60:197–204. doi: 10.1111/dgd.12537
21. Fleming DS, Miller LC. Differentially expressed miRNAs and tRNA genes affect host homeostasis during highly pathogenic porcine reproductive and respiratory syndrome virus infections in young pigs. Front Genet. (2019) 10:691. doi: 10.3389/fgene.2019.00691
22. Wright K, Plain K, Purdie A, Saunders BM, De Silva K. Biomarkers for detecting resilience against mycobacterial disease in animals. Infect Immun. (2020) 88:e00401-19 doi:10.1128/IAI.00401-19
23. Miretti S, Volpe MG, Martignani E, Accornero P, Baratta M. Temporal correlation between differentiation factor expression and microRNAs in Holstein bovine skeletal muscle. Animal. (2017) 11:227–35. doi: 10.1017/S1751731116001488
24. Erdos Z, Barnum JE, Wang E, Demaula C, Dey PM, Forest T, et al. Evaluation of the relative performance of pancreas-specific micrornas in rat plasma as biomarkers of pancreas injury. Toxicol Sci. (2020) 173:5–18. doi: 10.1093/toxsci/kfz184
25. Laterza OF, Lim L, Garrett-Engele PW, Vlasakova K, Muniappa N, Tanaka WK, et al. Plasma microRNAs as sensitive and specific biomarkers of tissue injury. Clin Chem. (2009) 55:1977–83. doi: 10.1373/clinchem.2009.131797
26. Lecchi C, Zamarian V, Gini C, Avanzini C, Polloni A, Rota Nodari S, et al. Salivary microRNAs are potential biomarkers for the accurate and precise identification of inflammatory response after tail docking and castration in piglets. J Anim Sci. (2020) 98:skaa153. doi: 10.1093/jas/skaa153
27. Samir M, Pessler F. Small non-coding RNAs associated with viral infectious diseases of veterinary importance: potential clinical applications. Front Vet Sci. (2016) 3:22. doi: 10.3389/fvets.201 6. 00022
28. Weber JA, Baxter DH, Zhang S, Huang DY, Huang KH, Lee MJ, et al. The microRNA spectrum in 12 body fluids. Clin Chem. (2010) 56:1733–41. doi: 10.1373/clinchem.2010.147405
29. Correia CN, Nalpas NC, McLoughlin KE, Browne JA, Gordon S V., MacHugh DE, et al. Circulating microRNAs as potential biomarkers of infectious disease. Front Immunol. (2017) 8:118. doi: 10.3389/fimmu.2017.00118
30. Lecchi C, Catozzi C, Zamarian V, Poggi G, Borriello G, Martucciello A, et al. Characterization of circulating miRNA signature in water buffaloes (Bubalus bubalis) during Brucella abortus infection and evaluation as potential biomarkers for non-invasive diagnosis in vaginal fluid. Sci Rep. (2019) 9:1945. doi: 10.1038/s41598-018-38365-x
31. Stenfeldt C, Arzt J, Smoliga G, Larocco M, Gutkoska J, Lawrence P. Proof-of-concept study: profile of circulating microRNAs in Bovine serum harvested during acute and persistent FMDV infection. Virol J. (2017) 14:71. doi: 10.1186/s12985-017-0743-3
32. Zhou C, Cai G, Meng F, Xu Z, He Y, Hu Q, et al. Deep-sequencing identification of MicroRNA biomarkers in serum exosomes for early pig pregnancy. Front Genet. (2020) 11:536. doi: 10.3389/fgene.2020.00536
33. Ioannidis J, Donadeu FX. Changes in circulating microRNA levels can be identified as early as day 8 of pregnancy in cattle. PLoS ONE. (2017) 12:e0174892. doi: 10.1371/journal.pone.0174892
34. Chen RJ, Kelly G, Sengupta A, Heydendael W, Nicholas B, Beltrami S, et al. MicroRNAs as biomarkers of resilience or vulnerability to stress. Neuroscience. (2015) 305:36–48. doi: 10.1016/j.neuroscience.2015.07.045
35. Xu YY, Xia QH, Xia QR, Zhang XL, Liang J. Microrna-based biomarkers in the diagnosis and monitoring of therapeutic response in patients with depression. Neuropsychiatr Dis Treat. (2019) 15:3583–97. doi: 10.2147/NDT.S237116
36. Jacobsen DP, Eriksen MB, Rajalingam D, Nymoen I, Nielsen MB, Einarsen S, et al. Exposure to workplace bullying, microRNAs and pain; evidence of a moderating effect of miR-30c rs928508 and miR-223 rs3848900. Stress. (2020) 23:77–86. doi: 10.1080/10253890.2019.1642320
37. Nordquist RE, van der Staay FJ, van Eerdenburg FJCM, Velkers FC, Fijn L, Arndt SS. Mutilating procedures, management practices, and housing conditions that may affect the welfare of farm animals: implications for welfare research. Animals. (2017) 7:12. doi: 10.3390/ani7020012
38. Carvalho RR, Palme R, da Silva Vasconcellos A. An integrated analysis of social stress in laying hens: the interaction between physiology, behaviour, and hierarchy. Behav Processes. (2018) 149:43–51. doi: 10.1016/j.beproc.2018.01.016
39. Proudfoot K, Habing G. Social stress as a cause of diseases in farm animals: current knowledge and future directions. Vet J. (2015) 206:15–21. doi: 10.1016/j.tvjl.2015.05.024
40. Koolhaas JM, Bartolomucci A, Buwalda B, de Boer SF, Flügge G, Korte SM, et al. Stress revisited: a critical evaluation of the stress concept. Neurosci Biobehav Rev. (2011) 35:1291–301. doi: 10.1016/j.neubiorev.2011.02.003
41. Reynolds RM. Glucocorticoid excess and the developmental origins of disease: two decades of testing the hypothesis – 2012 Curt Richter Award Winner. Psychoneuroendocrinology. (2013) 38:1–11. doi: 10.1016/j.psyneuen.2012.08.012
42. Wang Z, Gai Y, Zhou J, Liu J, Cui S. miR-375 mediates the CRF signaling pathway to regulate catecholamine biosynthesis by targeting Sp1 in porcine adrenal gland. Stress. (2019) 22:332–46. doi: 10.1080/10253890.2018.1561845
43. Ye RS, LiM, Li CY, Qi QE, Chen T, Cheng X, et al.MiR-361-3p regulates FSH by targeting FSHB in a porcine anterior pituitary cell model. Reproduction. (2017) 153:341–9. doi: 10.1530/REP-16-0373
44. He J, Zhang J, Wang Y, Liu W, Gou K, Liu Z, et al. MiR-7 mediates the zearalenone signaling pathway regulating FSH synthesis and secretion by targeting FOS in female pigs. Endocrinology. (2018) 159:2993-3006. doi: 10.1210/en.2018-00097
45. Qi QE, Xi QY, Ye RS, Chen T, Cheng X, Li CY, et al. Alteration of the miRNA expression profile in male porcine anterior pituitary cells in response to GHRH and CST and analysis of the potential roles for miRNAs in regulating GH. Growth Horm IGF Res. (2015) 25:66–74. doi: 10.1016/j.ghir.2014.1 2.002
46. Brogaard L, Heegaard PMH, Larsen LE, Mortensen S, Schlegel M, Dürrwald R, et al. Late regulation of immune genes and microRNAs in circulating leukocytes in a pig model of influenza A (H1N2) infection. Sci Rep. (2016) 6:21812. doi: 10.1038/srep21812
47. Brogaard L, Larsen LE, Heegaard PMH, Anthon C, Gorodkin J, Dürrwald R, et al. IFN-l and microRNAs are important modulators of the pulmonary innate immune response against influenza A (H1N2) infection in pigs. PLoS ONE. (2018) 13:e0194765. doi: 10.1371/journal.pone.0194765
48. Zhou M, Li C, Lu C, Zhang X, Pan Y, Liu X, et al. MiRNA29 promotes viral replication during early stage of PRRSV infection in vitro. DNA Cell Biol. (2016) 35:636–42. doi: 10.1089/dna.2015.3103
49. Zheng H, Xu L, Liu Y, Li C, Zhang L, Wang T, et al. MicroRNA-221-5p inhibits porcine epidemic diarrhea virus replication by targeting genomic viral RNA and activating the NF-kB pathway. Int J Mol Sci. (2018) 19:3381. doi: 10.3390/ijms19113381
50. Wu J, Ji Z, Qiao M, Peng X, Wu H, Song Z, et al. MicroRNA transcriptome analysis of poly I:C-stimulated and PRRSV-infected porcine alveolar macrophages. J Appl Genet. (2019) 60:375–83. doi: 10.1007/s13353-019-00500-3
51. Qi L, Wang K, Chen H, Liu X, Lv J, Hou S, et al. Host microRNA miR- 1307 suppresses foot-and-mouth disease virus replication by promoting VP3 degradation and enhancing innate immune response. Virology. (2019) 535:162–70. doi: 10.1016/j.virol.2019.07.009
52. Skovgaard K, Cirera S, Vasby D, Podolska A, Breum SO, Dürrwald R, et al. Expression of innate immune genes, proteins and microRNAs in lung tissue of pigs infected experimentally with influenza virus (H1N2). Innate Immun. (2013) 19:531–44. doi: 10.1177/1753425912473668
53. Jia X, Bi Y, Li J, Xie Q, Yang H, Liu W. Cellular microRNA miR-26a suppresses replication of porcine reproductive and respiratory syndrome virus by activating innate antiviral immunity. Sci Rep. (2015) 5:10651. doi: 10.1038/srep10651
54. Zhang Q, Guo XK, Gao L, Huang C, Li N, Jia X, et al. MicroRNA- 23 inhibits PRRSV replication by directly targeting PRRSV RNA and possibly by upregulating type I interferons. Virology. (2014) 450–51:182–95. doi: 10.1016/j.virol.2013.12.020
55. Liu D, Dai M, Zhang X, Cao W, Liao M. Subgroup J avian leukosis virus infection of chicken dendritic cells induces apoptosis via the aberrant expression of microRNAs. Sci Rep. (2016) 6:1–9. doi: 10.1038/srep20188
56. Núñez-Hernández F, Pérez LJ, Muñoz M, Vera G, Accensi F, Sánchez A, et al. Differential expression of porcine microRNAs in African swine fever virus infected pigs: a proof-of-concept study. Virol J. (2017) 14:198. doi: 10.1186/s12985-017-0864-8
57. Hata T, Murakami K, Nakatani H, Yamamoto Y, Matsuda T, Aoki N. Isolation of bovine milk-derived microvesicles carrying mRNAs and microRNAs. Biochem Biophys Res Commun. (2010) 396:528–33. doi: 10.1016/j.bbrc.2010.04.135
58. Gu Y, Li M, Wang T, Liang Y, Zhong Z, Wang X, et al. Lactation-related microRNA expression profiles of porcine breast milk exosomes. PLoS ONE. (2012) 7:e3691. doi: 10.1371/journal.pone.0043691
59. Chen T, Xi QY, Ye RS, Cheng X, Qi QE, Wang SB, et al. Exploration of microRNAs in porcine milk exosomes. BMC Genomics. (2014) 15:100. doi: 10.1186/1471-2164-15-100
60. Sun J, Zhang H, Hu B, Xie Y, Wang D, Zhang J, et al. Emerging roles of heat-induced circRNAs related to lactogenesis in lactating sows. Front Genet. (2020) 10:1347. doi: 10.3389/fgene.2019.01347
61. Xie MY, Chen T, Xi QY, Hou LJ, Luo JY, Zeng B, et al. Porcine milk exosome miRNAs protect intestinal epithelial cells against deoxynivalenol-induced damage. Biochem Pharmacol. (2020) 175:113898. doi: 10.1016/j.bcp.2020.113898
62. Xie MY, Hou LJ, Sun JJ, Zeng B, Xi QY, Luo JY, et al. Porcine milk exosome MiRNAs attenuate LPS-induced apoptosis through inhibiting TLR4/NF-kB and p53 pathways in intestinal epithelial cells. J Agric Food Chem. (2019) 67:9477–91. doi: 10.1021/acs.jafc.9b02925
63. Tao X, Xu Z. MicroRNA transcriptome in swine small intestine during weaning stress. PLoS ONE. (2013) 8:e79343. doi: 10.1371/journal.pone.0079343
64. Wang X, You Z, Zhao G, Wang T. MicroRNA-194-5p levels decrease during deep hypothermic circulatory arrest. Sci Rep. (2018) 8:14044. doi: 10.1038/s41598-018-32426-x
65. Enserink JM. Sumo and the cellular stress response. Cell Div. (2015) 10:4. doi: 10.1186/s13008-015-0010-1
66. Mount LE. Adaptation to Thermal Environment. Man and His Productive Animals. London: Edward Arnold Publishers Ltd. (1979).
67. Brown-Brandl TM, Nienaber JA, Xin H, Gates RS. A literature review of swine heat and moisture. Trans ASAE. (2004) 47:259–70. doi: 10.13031/2013.15867
68. Amavizca-Nazar A, Montalvo-Corral M, González-Rios H, Pinelli-Saavedra A. Hot environment on reproductive performance, immunoglobulins, vitamin E, and vitamin A status in sows and their progeny under commercial husbandry. J Anim Sci Technol. (2019) 61:340–51. doi: 10.5187/jast.2019.61.6.340
69. Renaudeau D, Quiniou N, Noblet J. Effects of exposure to high ambient temperature and dietary protein level on performance of multiparous lactating sows. J Anim Sci. (2001) 79:1240–9. doi: 10.2527/2001.7951240x
70. Lucy MC, Safranski TJ. Heat stress in pregnant sows: thermal responses and subsequent performance of sows and their offspring.Mol Reprod Dev. (2017) 84:946–56. doi: 10.1002/mrd.22844
71. Huo C, Xiao C, She R, Liu T, Tian J, Dong H, et al. Chronic heat stress negatively affects the immune functions of both spleens and intestinal mucosal system in pigs through the inhibition of apoptosis. Microb Pathog. (2019) 136:103672. doi: 10.1016/j.micpath.2019.103672
72. Quiniou N, Noblet J. Influence of high ambient temperatures on performance of multiparous lactating sows. J Anim Sci. (1999) 77:2124–34. doi: 10.2527/1999.7782124x
73. Kamanga-Sollo E, Pampusch MS, White ME, Hathaway MR, Dayton WR. Effects of heat stress on proliferation, protein turnover, and abundance of heat shock protein messenger ribonucleic acid in cultured porcine muscle satellite cells. J Anim Sci. (2011) 89:3473–80. doi: 10.2527/jas.2011-4123
74. Hao Y, Liu JR, Zhang Y, Yang PG, Feng YJ, Cui YJ, et al. The microRNA expression profile in porcine skeletal muscle is changed by constant heat stress. Anim Genet. (2016) 47:365–9. doi: 10.1111/age.12419
Te puede interesar:
La programación fetal y su impacto sobre los marcadores epigenéticos de la inmunidad de los lechones
[/registrados]
