LA TÉCNICA MALDI-TOF APORTA RAPIDEZ, OBJETIVIDAD Y PRECISIÓN EN LA IDENTIFICACIÓN DE PATÓGENOS
Para leer más contenidos de Revista porciSapiens Abril 2022
Para leer más contenidos de Revista porciSapiens Abril 2022
| El diagnóstico de laboratorio de enfermedades infecciosas tiene como objetivo identificar el agente o los agentes etiológicos causantes de un proceso clínico. Esta información permite instaurar un tratamiento que sea eficaz, así como aplicar medidas de prevención para evitar nuevos brotes.
La presencia de un agente infeccioso puede determinarse de forma directa detectando el agente en la muestra clínica tomada del animal, por ejemplo, mediante aislamiento microbiológico o detección de su genoma mediante técnicas como la reacción en cadena de la polimerasa (PCR). Sin embargo, también es posible hacerlo de una forma indirecta, utilizando las técnicas serológicas que detectan los anticuerpos generados por el animal frente al patógeno. |

| En este artículo se repasan las novedades recientes a nivel de diagnóstico de laboratorio. Estas nuevas tecnologías han aportado grandes ventajas tanto en rapidez como en sensibilidad frente a las clásicas. |
NUEVAS TÉCNICAS Y TECNOLOGÍAS
CULTIVO MICROBIOLÓGICO Y SUSCEPTIBILIDAD A ANTIMICROBIANOS
![]()
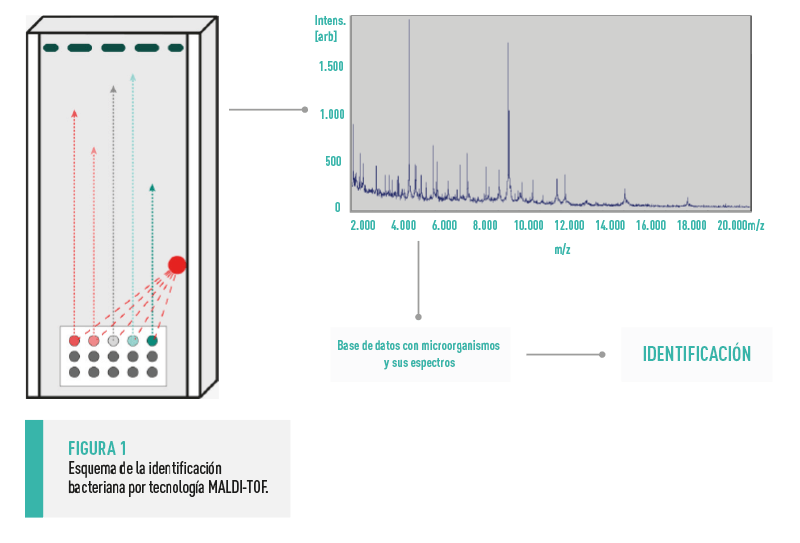
En el laboratorio de microbiología, el uso de la tecnología de espectrometría de masas MALDI-TOF (de sus siglas en inglés Matrix-Assisted Laser Desorption/Ionization Time of Flight) ha ido reemplazando rápidamente a las técnicas de identifi cación bioquímicas. Esta tecnología es capaz de identificar microorganismos a través de su conjunto de proteínas (poseen una huella molecular específica de especie).
![]() La colonia aislada se introduce en [registrados]el equipo donde se rompe por acción de un láser y todas sus proteínas se detectan generando un espectro característico. Comparando este espectro con una base de datos con miles de patógenos y sus respectivos espectros somos capaces de conocer qué microorganismo es en cuestión de minutos (Figura 1).
La colonia aislada se introduce en [registrados]el equipo donde se rompe por acción de un láser y todas sus proteínas se detectan generando un espectro característico. Comparando este espectro con una base de datos con miles de patógenos y sus respectivos espectros somos capaces de conocer qué microorganismo es en cuestión de minutos (Figura 1).
LA TÉCNICA MALDI-TOF APORTA RAPIDEZ, OBJETIVIDAD Y PRECISIÓN EN LA IDENTIFICACIÓN DE PATÓGENOS
Por ejemplo, en el caso del género Brachyspira, aquellas especies de hemólisis débil ahora se identifican fácilmente. Otro ejemplo lo encontramos en los Enterococcus digestivos o los Staphylococcus coagulasa negativos, que actualmente se están identificando a nivel de especie, lo que permite acumular datos y detectar patógenos emergentes.
PRUEBAS DE SENSIBILIDAD ANTIBIÓTICA
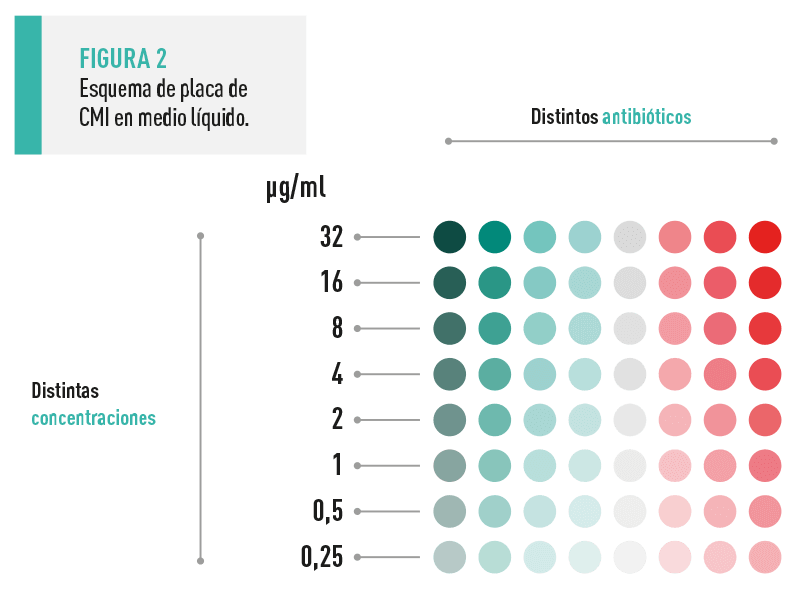
Cuando un agente bacteriano es identificado y considerado implicado en el proceso clínico, el siguiente paso es realizar pruebas de sensibilidad antibiótica para instaurar un tratamiento.
La aparición de resistencias es un problema actual en la salud pública. España está en marcha el plan nacional frente a la resistencia a antibióticos para controlar su uso y evitar la expansión de dichas resistencias tanto en el ámbito veterinario como humano.
La técnica más extendida para este propósito es el método disco-placa o Kirby-Bauer, donde una única concentración de antibiótico se enfrenta a la bacteria para observar la posible inhibición de su crecimiento.

TÉCNICAS DE BIOLOGÍA MOLECUL AR
![]()
PCR A TIEMPO REAL
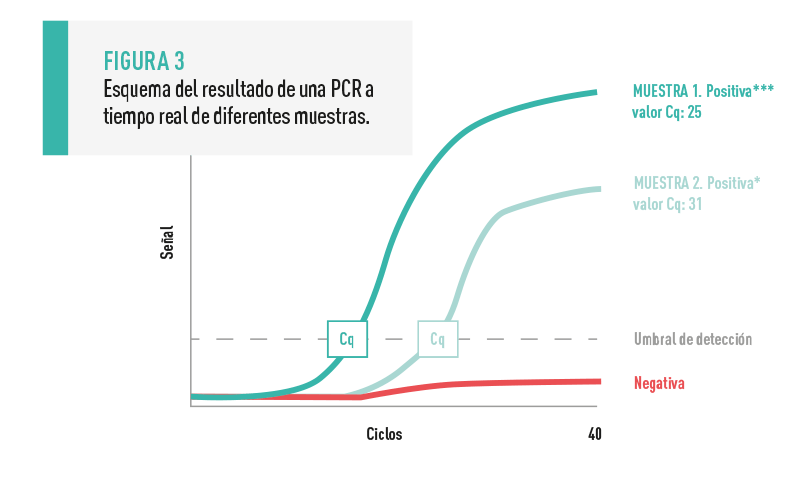
En cuanto a las técnicas moleculares de diagnóstico, cabe destacar la PCR a tiempo real o PCR cuantitativa que, si bien puede no considerarse novedad, ya que se emplea hace más de una década, actualmente sigue siendo la técnica de referencia y más extendida para el diagnóstico de muchas enfermedades infecciosas.
Las diferentes especies patógenas, así como serotipos, toxinas o factores de virulencia con papeles clave en un proceso clínico están codificados por secuencias genómicas únicas.
| A diferencia de la PCR convencional, la PCR a tiempo real es una técnica con mayor sensibilidad y especificidad en la que el fragmento de genoma amplificado se detecta tras cada ciclo de amplificación, lo que posibilita una cuantificación del agente en la muestra inicial (Figura 3).
|
SECUENCIACIÓN
SECUENCIACIÓN DE SANGER
En muchos casos, saber qué microorganismo está presente no es suficiente para tomar las medidas adecuadas de prevención. En este sentido, la secuenciación de ADN, que consiste en la determinación de los nucleótidos de un gen o región genómica, puede ser de gran utilidad.
La técnica empleada en estos casos es la secuenciación de Sanger y, una vez obtenida la secuencia, su análisis y comparación con otras secuencias nos permite obtener información diversa.
![]() Por ejemplo, en el caso del virus PRRS, la secuenciación nos proporciona información epidemiológica. En el caso del rotavirus A o la influenza A, también podemos conocer el genotipo contrastando las secuencias de dos de sus genes con las bases de datos disponibles, por ejemplo: Rotavirus G(9)/P[23] o Influenza H1 aviar/N2.
Por ejemplo, en el caso del virus PRRS, la secuenciación nos proporciona información epidemiológica. En el caso del rotavirus A o la influenza A, también podemos conocer el genotipo contrastando las secuencias de dos de sus genes con las bases de datos disponibles, por ejemplo: Rotavirus G(9)/P[23] o Influenza H1 aviar/N2.
Conocer la secuencia de la cepa circulante o su genotipo puede dar información acerca del origen de un brote:
![]() Podremos establecer si ha existido un problema de bioseguridad externa, dónde se ha podido producir una entrada lateral y la secuencia obtenida para esa cepa que no habíamos encontrado antes en esa explotación.
Podremos establecer si ha existido un problema de bioseguridad externa, dónde se ha podido producir una entrada lateral y la secuencia obtenida para esa cepa que no habíamos encontrado antes en esa explotación.
![]() Si volvemos a encontrar una cepa con una alta similitud a las cepas previamente detectadas, la solución implicará mejoras a nivel de bioseguridad interna.
Si volvemos a encontrar una cepa con una alta similitud a las cepas previamente detectadas, la solución implicará mejoras a nivel de bioseguridad interna.
Por otro lado, se puede determinar si se está detectando la cepa vacunal en los casos en los que la vacunación se realiza con cepas vivas atenuadas, lo que descartaría fallos de bioseguridad y la implicación de ese agente en la clínica de los animales.
TÉCNICA MLST
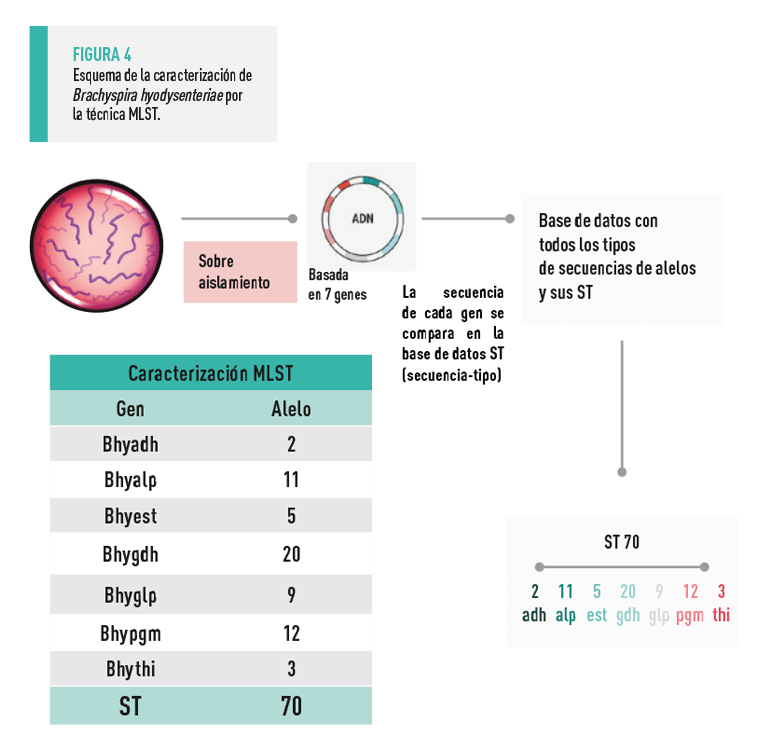
La secuenciación también puede aplicarse para diferenciar entre cepas de bacterias. La metodología MLST (de las siglas en inglés, Multiple Locus Sequence Typing) consiste en secuenciar 7 genes de la bacteria.
![]() La secuencia de cada uno de ellos es asignada a un número de alelo en función de los nucleótidos que contiene cada gen. A su vez, la combinación de los números de alelos de los 7 genes da una secuencia tipo o ST (de sus siglas en inglés, Sequence Type) (Figura 4). Por lo tanto, dos cepas se considerarán diferentes si pertenecen a dos ST distintos. En porcino, cabe destacar su uso en el estudio de brotes de disentería porcina para esclarecer el origen epidemiológico tras una erradicación previa.
La secuencia de cada uno de ellos es asignada a un número de alelo en función de los nucleótidos que contiene cada gen. A su vez, la combinación de los números de alelos de los 7 genes da una secuencia tipo o ST (de sus siglas en inglés, Sequence Type) (Figura 4). Por lo tanto, dos cepas se considerarán diferentes si pertenecen a dos ST distintos. En porcino, cabe destacar su uso en el estudio de brotes de disentería porcina para esclarecer el origen epidemiológico tras una erradicación previa.
ANÁLISIS MLVA
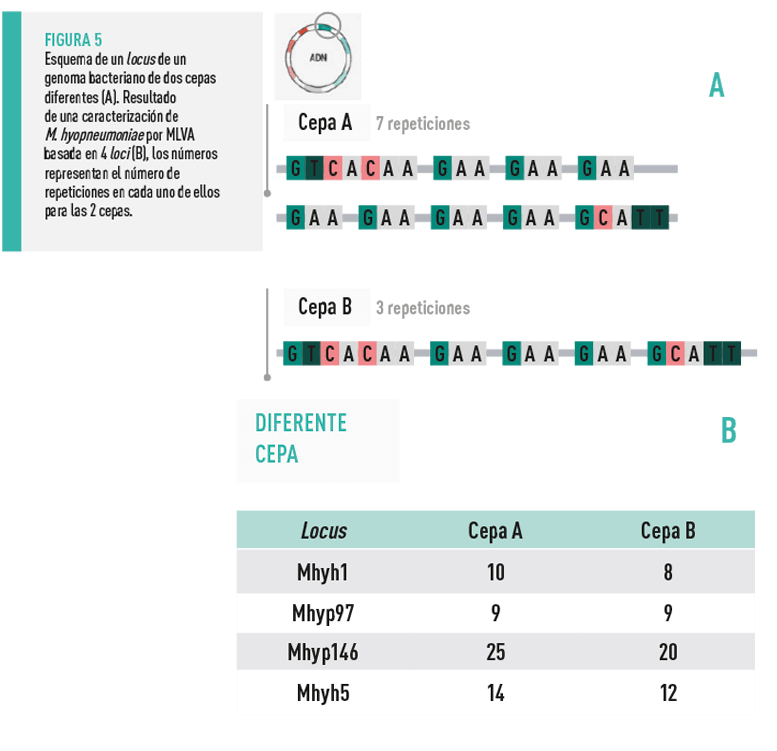
Otra técnica, que en porcino se ha descrito para Mycoplasma hyopneumoniae es la de Multi Locus VNTR Analysis (MLVA), donde VNTR viene de Variable Number Tandem Repeats. Las bacterias tienen en su genoma diferentes puntos (locus) que contienen grupos de nucleótidos repetidos un número determinado de veces (repeticiones variables en tándem).
![]() Cepas aisladas en distintos brotes con el mismo número de repeticiones en tándem en cada uno de estos locus pueden considerarse una misma cepa, y al contrario, si se encuentran variaciones, se puede establecer que son diferentes.
Cepas aisladas en distintos brotes con el mismo número de repeticiones en tándem en cada uno de estos locus pueden considerarse una misma cepa, y al contrario, si se encuentran variaciones, se puede establecer que son diferentes.
Al secuenciar estos loci (plural de locus) de varias cepas podemos saber cuántas repeticiones hay de cada grupo de nucleótidos y determinar si son la misma o diferentes (Figura 5).
PERSPECTIVAS DE FUTURO
Las técnicas moleculares en las que se analizan ácidos nucleicos presentan un futuro prometedor en el diagnóstico de enfermedades infecciosas en veterinaria.
En la última década se han puesto a punto nuevas formas de secuenciar el ADN (diferentes a la técnica de Sanger), denominadas técnicas de secuenciación de nueva generación y de tercera generación. También se conocen como técnicas de secuenciación masiva, ya que se generan una gran cantidad de secuencias.

Aunque presenta ventajas frente a Sanger y proporciona gran cantidad de información, el coste de realizarla es todavía muy elevado y el análisis e interpretación es de gran complejidad.
![]() Por el momento, toda esta cantidad de nuevas secuencias aportadas a las bases de datos es clave, pues sirve como referencia para poder desarrollar y mejorar las técnicas mencionadas anteriormente (PCR a tiempo real y secuenciación Sanger) ya que para su aplicación se requiere el conocimiento previo del genoma de los agentes a estudiar.
Por el momento, toda esta cantidad de nuevas secuencias aportadas a las bases de datos es clave, pues sirve como referencia para poder desarrollar y mejorar las técnicas mencionadas anteriormente (PCR a tiempo real y secuenciación Sanger) ya que para su aplicación se requiere el conocimiento previo del genoma de los agentes a estudiar.
Por otro lado, hay técnicas en proceso de desarrollo que permitirían un diagnóstico in situ, a pie de campo. Una de ellas es la PCR isoterma, una variante de la PCR que no requiere de equipamiento para su realización al realizarse a una temperatura constante.
![]() Se han descrito diferentes metodologías donde se acopla esta reacción de PCR a un sistema de detección a la vista como una tira reactiva o la observación de un cambio de color en el tubo cuando la reacción tiene un resultado positivo.
Se han descrito diferentes metodologías donde se acopla esta reacción de PCR a un sistema de detección a la vista como una tira reactiva o la observación de un cambio de color en el tubo cuando la reacción tiene un resultado positivo.
Otra metodología que se puede aplicar en el diagnóstico in situ es la de CRISPR. Se trata de un complejo de proteínas capaces de reconocer y editar regiones específicas de un genoma. Sin embargo, existe una versión de CRISPR que identifica las regiones genómicas de interés y, cuando esto ocurre, el complejo se activa, provocando una rotura de una molécula fluorescente, revelando un positivo y la presencia del patógeno de estudio en la muestra (Figura 6).
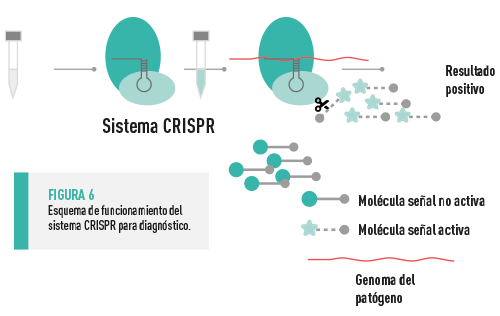
Ambas tecnologías han sido descritas para la detección de PRRSV en el campo y presentan una buena sensibilidad y especificidad, con facilidad de uso y sin necesidad de equipamiento.

| Existe una gran variedad de técnicas para realizar el diagnóstico de una enfermedad infecciosa y cada vez se desarrollan nuevas tecnologías. Sin embargo, es importante conocerlas y elegir la adecuada para responder a las preguntas que se plantea el veterinario.
|
BIBLIOGRAFÍA
Chang Y, Deng Y, Li T, Wang J, Wang T, Tan F, Li X, Tian K. Visual detection of porcine reproductive and respiratory syndrome virus using CRISPR-Cas13a. Transbound Emerg Dis. 2020 Mar;67(2):564-571. doi: 10.1111/tbed.13368. Epub 2019 Sep 30. PMID: 31541593.
Finlay BJ, Esteban GF. Exploring Leeuwenhoek’s legacy: the abundance and diversity of protozoa. Int Microbiol. 2001 Sep;4(3):125-33. doi: 10.1007/s10123- 001-0027-y. PMID: 11820430.
La T, Phillips ND, Harland BL, Wanchanthuek P, Bellgard MI, Hampson DJ. Multilocus sequence typing as a tool for studying the molecular epidemiology and population structure of Brachyspira hyodysenteriae. Vet Microbiol. 2009 Sep 18;138(3-4):330-8. doi: 10.1016/j.vetmic.2009.03.025. Epub 2009 Mar 25. PMID: 19369014.
Park JY, Park S, Park YR, Kang DY, Kim EM, Jeon HS, Kim JJ, Kim WI, Lee KT, Kim SH, Lee KK, Park CK. Reverse-transcription loop-mediated isothermal amplification (RT-LAMP) assay for the visual detection of European and North American porcine reproductive and respiratory syndrome viruses. J Virol Methods. 2016 Nov;237:10-13. doi: 10.1016/j. jviromet.2016.08.008. Epub 2016 Aug 16. PMID: 27542532.
Prohaska S, Pflüger V, Ziegler D, Scherrer S, Frei D, Lehmann A, Wittenbrink MM, Huber H. MALDI-TOF MS for identification of porcine Brachyspira species. Lett Appl Microbiol. 2014 Mar;58(3):292-8. doi: 10.1111/lam.12189. Epub 2013 Nov 18. PMID: 24236892.
Van Borm S, Belák S, Freimanis G, Fusaro A, Granberg F, Höper D, King DP, Monne I, Orton R, Rosseel T. Next-generation sequencing in veterinary medicine: how can the massive amount of information arising from high-throughput technologies improve diagnosis, control, and management of infectious diseases? Methods Mol Biol. 2015;1247:415-36. doi: 10.1007/978-1-4939-2004-4_30. PMID: 25399113; PMCID: PMC7123048.
Vranckx K, Maes D, Calus D, Villarreal I, Pasmans F, Haesebrouck F. Multiple-locus variable number tandem-repeat analysis is a suitable tool for differentiation of Mycoplasma hyopneumoniae strains without cultivation. J Clin Microbiol. 2011 May;49(5):2020-3. doi: 10.1128/JCM.00125-11. Epub 2011 Mar 9. PMID: 21389157; PMCID: PMC3122671.
[/registrados]
