Es muy importante establecer las pautas de muestreo para diagnosticar y monitorizar cada tipo de estas situaciones en el campo.
El diagnóstico de la gripe porcina requiere establecer unas pautas de muestreo para responder rápidamente a la aparición del virus en las explotaciones.

En ocasiones es difícil detectar el virus, en especial en las explotaciones donde se muestra la forma enzoótica de la enfermedad.
Es muy importante establecer las pautas de muestreo para diagnosticar y monitorizar cada tipo de estas situaciones en el campo.
Gripe porcina – El virus
El virus de la gripe porcina causa una infección aguda en el aparato respiratorio.
La duración de la infección se estima entre 5 y 7 días desde su inicio, con picos de carga vírica entre el tercer y quinto día de infección. Esto hace que sea muy complicado poder detectar a tiempo al virus en un animal infectado.
Para poder abordar el diagnóstico de esta enfermedad es importante contar con un plan de actuación en la mayor brevedad posible desde la aparición de los signos clínicos, si los hay.
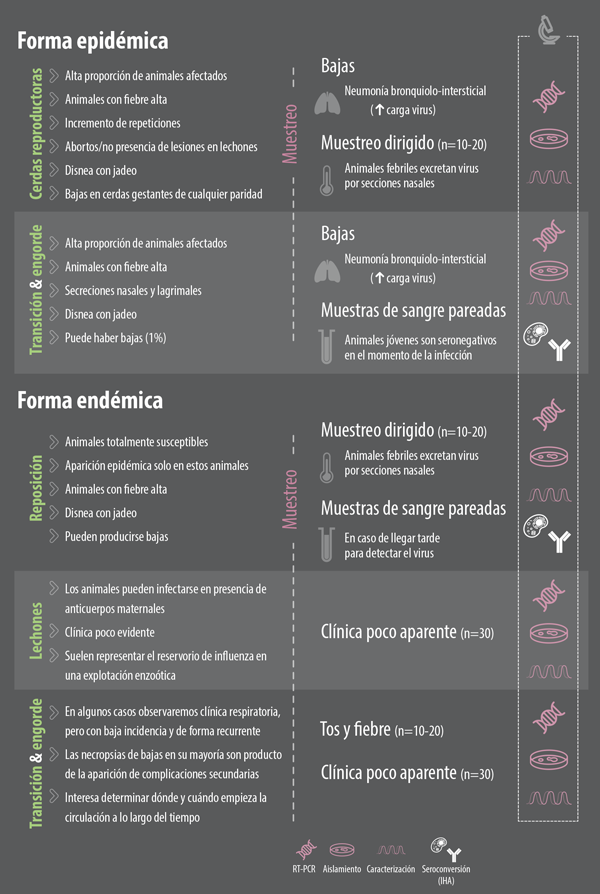
Un primer aspecto que se debe tener en cuenta para el diagnóstico de la enfermedad es saber frente a qué forma epidemiológica nos encontramos.
En la actualidad, la gripe porcina se encuentra básicamente de dos formas en las explotaciones porcinas:
FORMA EPIDÉMICA
La forma epidémica suele producirse cuando se introduce una nueva cepa del virus en una explotación para la cual no existe inmunidad previa en la población.
Ante esta situación nos encontraremos con animales infectados en cualquier fase en la granja, y nos llamará especialmente la atención:
La neumonía bronquiolo-intersticial es la misma lesión que causa Mycoplasma hyopneumoniae, por lo que hay que tenerlo en cuenta en el diagnóstico diferencial.
Es recomendable enviar los pulmones al laboratorio, ya que el virus suele detectarse (por RT-PCR) o aislarse (en cultivo celular o huevo embrionado de pollo) fácilmente en caso de estar presente.
Otra técnica que puede realizarse a partir de los pulmones es la inmunohistoquímica, que tiene la ventaja de poder asociar la presencia de virus con la lesión a nivel microscópico, pero no es tan práctica y rápida como la RT-PCR desde un punto de vista de diagnóstico.
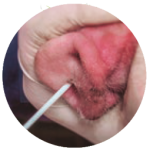
En caso de no encontrar animales RT-PCR positivos (muestreo demasiado tarde) puede ser recomendable obtener muestras pareadas en animales jóvenes (animales de transición a 6-7 semanas de vida/cerdas de reposición de origen externo negativo) (Foto 1) que acaben de sufrir el cuadro clínico y 21 días después.

Foto 1. Se pueden obtener muestras en animales de transición que acaben de sufrir el cuadro clínico
[registrados]
Obtener muestras pareadas en animales jóvenes nos permitirá determinar si existe seroconversión frente al virus de la gripe ya sea de forma genérica al tipo A (por ELISA) o específica a subtipo (por inhibición de la hemaglutinación)
Las muestras pareadas en cerdas multíparas o en animales de engorde no suelen aportar demasiada información, ya que el 75% y 50% de estos animales ya son seropositivos, respectivamente.
FORMA ENZOÓTICA
En aquellas situaciones en las que existe cierta inmunidad previa en la población, el virus de la gripe suele establecer un ciclo enzoótico de infección con menor incidencia que en la forma endémica.
Ante esta situación, el virus suele mantenerse en ciclos de infección recurrentes en animales susceptibles.
Por tanto, el muestreo de estas subpoblaciones de animales susceptibles será fundamental en esta forma.
A día de hoy se considera que el principal reservorio del virus en una explotación endémica son los animales en maternidad. Ello se debe al hecho de que los anticuerpos maternales protegen ante la clínica pero no ante la infección.
Por tanto, una primera aproximación diagnóstica podría ser realizar un muestreo de hisopos nasales en esta fase en función de una prevalencia esperada (por ejemplo, 30 hisopos permiten detectar una prevalencia del 10% como mínimo con un 95% de confianza) para analizarlos por RT-PCR.
La interferencia con anticuerpos maternales hace que un animal infectado en esta fase no desarrolle anticuerpos frente al virus, por lo que las muestras pareadas no son de utilidad.
En otras fases, como la transición o el engorde, la desaparición de anticuerpos maternales deja a los animales susceptibles a la infección, de ahí que aparezcan cuadros respiratorios de tipo “recurrente” en estos animales.
Si la clínica es evidente (tos seca o fiebre), es recomendable realizar un muestreo dirigido mediante hisopos nasales.
En esta fase, las bajas suelen deberse a complicaciones bacterianas secundarias, por lo que la necropsia solo nos indicará cuáles son las infecciones concomitantes presentes en la explotación pero no tendrán un valor diagnóstico en cuanto a gripe.
Si no observamos clínica compatible o evidente o solamente observamos complicaciones bacterianas, otra aproximación posible es la monitorización periódica mediante fluidos orales, lo que nos permitirá determinar dónde y cuándo circula el virus.
Al tratarse de una muestra agregada (obtenida a partir de varios animales), no debería utilizarse para realizar la caracterización del virus.
En caso de que se desee, es recomendable determinar qué corrales son positivos mediante cuerdas y volver a muestrear individualmente los animales mediante hisopos nasales en una proporción determinada de los animales presentes en el corral al cabo de pocos días (2-3 días).
Población de reposición
Otra población que hay que tener muy en cuenta ante la forma enzoótica es la reposición, especialmente en aquellos casos en los que llegue de un origen negativo externo (Foto 2).

Foto 2. Frente a la forma enzoótica hay que tener muy en cuenta a la población de reposición
Si la adaptación se realiza dentro de la explotación sin previa inmunización, estos animales pueden llegar a sufrir un episodio epidémico a pequeña escala. En caso de que ello se produzca, es recomendable aplicar las pautas descritas en la forma epidémica.
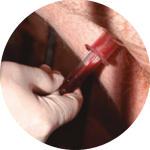
Foto 3. Las muestras de sangre permiten evaluar la seroconversión
En estos animales puede ser también de gran utilidad obtener muestras pareadas de sangre con el fin de evaluar la seroconversión, ya que con total seguridad partirán de la ausencia de anticuerpos a títulos elevados debidos a la infección (Foto 3).
Finalmente, hay que insistir en que para el aislamiento y la caracterización (ya sea mediante secuenciación o determinación del subtipo o el linaje de hemaglutinina o neuraminidasa por RT-PCR) es imprescindible trabajar a partir de muestras individuales, ya que en ocasiones podemos detectar más de una cepa de virus de la gripe en la misma población, lo que imposibilita la interpretación de resultados a partir de muestras agregadas o pooles.
Proyecto de investigación
Actualmente, el Departamento de Sanidad y de Anatomía Animal de la Universidad Autónoma de Barcelona e IDT Biologika han iniciado un proyecto de investigación con el fin de determinar la diversidad genética de los virus de la gripe porcina presentes a día de hoy en la cabaña porcina de la península ibérica, así como estudiar la epidemiología y el impacto de estos virus frente a distintos escenarios o formas de la enfermedad.
Con ese fin se ha iniciado la búsqueda activa de casos tanto de gripe epidémica como enzoótica en explotaciones de porcino de esta zona geográfica.
Como ya se ha comentado en este artículo, en determinadas ocasiones es difícil llegar a detectar el virus, especialmente en aquellas explotaciones donde se observa la enfermedad en su forma enzoótica sin presencia de una clínica compatible aparentemente.
Es muy importante establecer y normalizar las pautas de muestreo para poder diagnosticar o monitorizar cada tipo de estas situaciones en el campo
Para colaborar con este proyecto, y enviar muestras de casos donde sospeche de Influenza y quiera obtener más información, gracias por contactar a:
PROYECTO INFLUENZA
Lab. Infeccioses – Fac. Veterinaria Campus UAB
08193 – Cerdanyola del Vallès
935 812 807
[registrados]
Suscribete ahora a la revista técnica porcina
AUTORES
Cómo cuidar la salud respiratoria de los cerdos: medidas clave
Alejandro Cid González Ana I. Pastor Calonge César B. Gutiérrez Martín Mario Delgado García Óscar Mencía-Ares Sonia Martínez Martínez
2026: consolidar fortalezas para avanzar con firmeza

Respuesta de emergencia en accidentes con transporte porcino: la experiencia danesa
Cecilie Kobek-Kjeldager Kirstin Dahl-Pedersen Mette S. Herskin
Microbiota intestinal en el cerdo: de la taxonomía a la función en producción porcina
Eduard de la Torre Candán
Energía neta y crecimiento porcino: claves para una mejor predicción
Gabriela Martínez
La apuesta ganadora de Porgaor, Grupo LANKA: el sistema de alimentación individualizada en maternidades, Materneo de Asserva

El arte de alimentar a cerdas reproductoras: inteligencia artificial al servicio de la nutrición y productividad
Gustavo Márquez Esteban Pablo Fuentes Pardo Sara Crespo Vicente
Lechones más fuertes y numerosos al destete

Estrategia nutricional active feeding en lechones destetados PRRS positivos
Alberto Morillo Alujas
MASTERFLY® BAIT: control eficaz y duradero de moscas

El destete como punto clave de la productividad: puntos críticos a tener en cuenta
Anabel Fernández Bravo Andrea Martínez Martínez Elena Goyena Salgado Emilio José Ruiz Fernández Francisco Frances Diaz José Manuel Pinto Carrasco Manuel Toledo Castillo Simón García Legaz
Medidas para el control de la cepa Rosalía de PRRS

Gestionar la incertidumbre cuando el miedo entra en la granja
Laura Pérez Sala
Bioseguridad y control: estrategias para afrontar el PRRS y PPA en un escenario de riesgo

Bioseguridad aplicada frente a Peste Porcina Africana: protocolos clave en granja, logística y control externo
David García Páez