Diagnóstico molecular del PRRS: de la PCR a tiempo real a la secuenciación masiva
Para leer más contenidos de Revista porciSapiens Abril 2023
Para leer más contenidos de Revista porciSapiens Abril 2023
| El Síndrome Reproductivo y Respiratorio Porcino (PRRS) es una de las enfermedades más importantes en la industria porcina a nivel mundial ya que ocasiona elevadas pérdidas económicas1,2. Este impacto económico está asociado principalmente al aumento de la mortalidad en lechones, la necesidad del uso de vacunas y la implementación de medidas de bioseguridad3. La enfermedad afecta a cerdas reproductoras, lechones y cerdos jóvenes en crecimiento.
En cerdas se observan problemas reproductivos como abortos, repeticiones, mayor incidencia de mortinatos, fetos momificados y lechones nacidos débiles. Además, puede ocasionar disminución en el crecimiento y aumento de la tasa de mortalidad en lechones por problemas respiratorios y susceptibilidad a otros agentes patógenos4. |
EL VIRUS PRRS
Entre ambos genotipos existen grandes diferencias a nivel de genoma, ya que solo comparten entre un 50-70% de identidad nucleotídica.
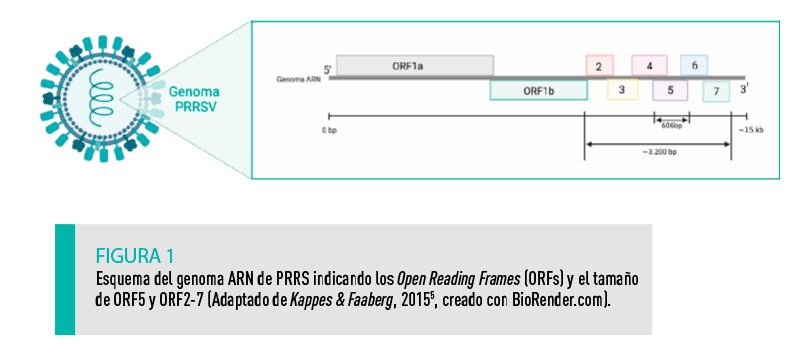
| Dentro de un mismo genotipo existe gran variabilidad entre cepas, pudiendo llegar a diferir en hasta el 15% en el genoma6,7. El tamaño del genoma del PRRSV es de unos 15.000 pares de bases, correspondiendo tres cuartas partes al ORF1, que codifica para proteínas no estructurales. El resto del genoma comprende el resto de ORF (ORF2a, ORF2b, ORF3, ORF4, ORF5, ORF6 y ORF7) que codifican para las proteínas estructurales del virus (Figura 1)8. |
LA GRAN VARIABILIDAD GENÉTICA DEL PRRSV ES FRUTO DE SU ELEVADA TASA DE MUTACIÓN Y DE LOS FENÓMENOS DE RECOMBINACIÓN
En un animal infectado, lo habitual es que se encuentre un conjunto de cuasiespecies virales sobre las que actúa la presión selectiva derivada de la inmunidad del hospedador9,10. Todo ello ha conducido al extenso abanico de cepas circulantes PRRS en todo el mundo11.

DIAGNÓSTICO DE PRRS
El grave impacto productivo derivado del PRRS ha hecho que, desde su descubrimiento a finales de los años ochenta, el virus siga siendo objeto de numerosas investigaciones con el objetivo de entender los mecanismos que intervienen en su patogénesis y epidemiología, así como desarrollar estrategias para el control efectivo de la enfermedad.

| Existen diferentes herramientas de diagnóstico disponibles actualmente. A continuación, repasaremos las principales técnicas empleadas actualmente, incidiendo en su utilidad práctica. |
TÉCNICAS INMUNOLÓGICAS Y CULTIVO VIRAL
El uso de técnicas serológicas es común para la detección de anticuerpos frente al virus, aunque no permiten diferenciar anticuerpos de infecciones activas de los producidos como respuesta a la vacunación.

Por otro lado, a pesar de la complejidad y el largo tiempo de análisis, el aislamiento y cultivo viral también se han empleado como método de diagnóstico. Además, técnicas como la inmunohistoquímica y la inmunofluorescencia permiten detectar el antígeno viral pero su sensibilidad es limitada14.
TÉCNICAS MOLECULARES
Las técnicas moleculares basadas en la detección del genoma viral, como la reacción en cadena de la polimerasa (PCR), confieren gran sensibilidad y especificidad15.

LA PCR ES LA HERRAMIENTA MÁS UTILIZADA EN LA ACTUALIDAD Y PERMITE ANALIZAR VARIOS TIPOS DE MUESTRAS DIFERENTES
La elección del tipo de muestra dependerá del objetivo del análisis:
Confirmación de una sospecha clínica
|
Monitorización del virus para conocer el estado epidemiológico de la explotación
*El uso de estas últimas cada |
PCR A TIEMPO REAL
![]()
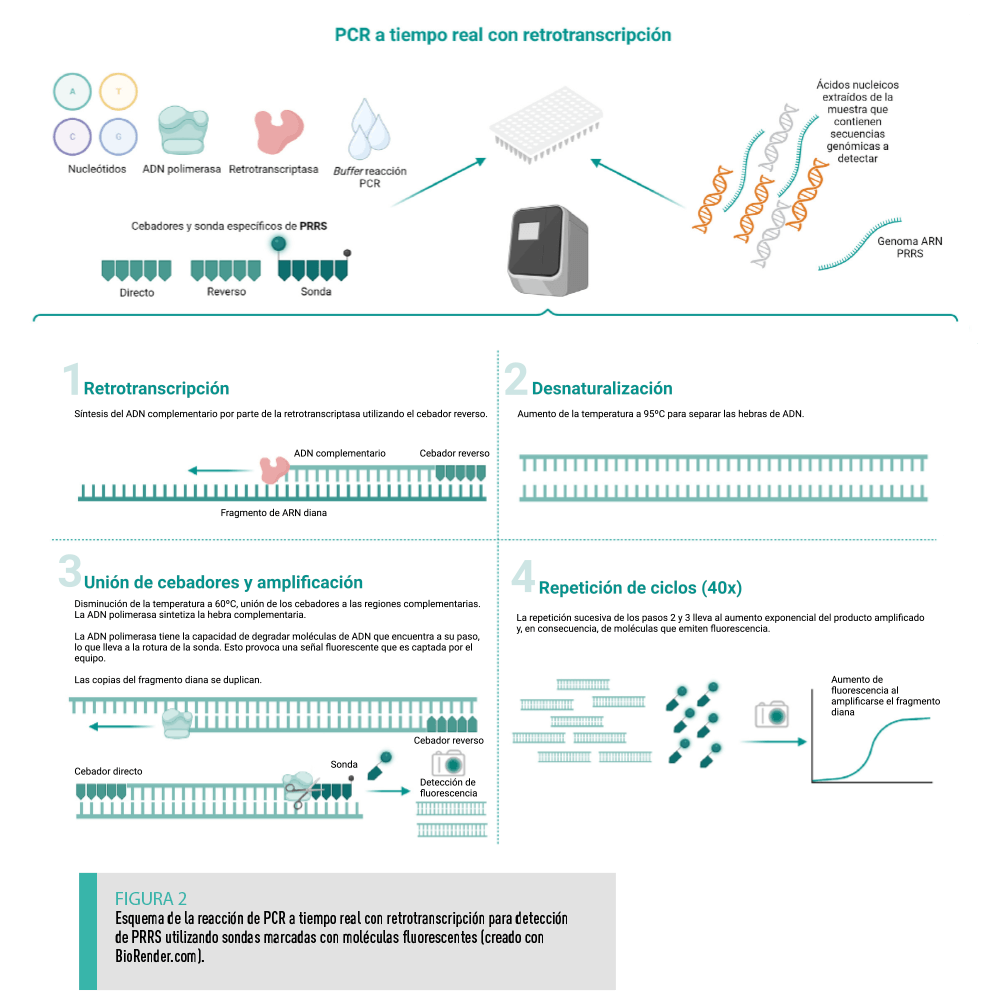
La PCR a tiempo real o cuantitativa (qPCR) es actualmente la técnica más extendida entre los laboratorios veterinarios para realizar el diagnóstico y monitorización de patógenos, incluido el PRRSV.
Aunque la PCR convencional con retrotranscripción o transcripción inversa (RT-PCR) ya se utilizaba para el diagnóstico de PRRSV21, la qPCR, llevando a cabo el paso previo necesario de retrotranscripción del genoma ARN del virus (RT-qPCR), ha terminado por reemplazarla, ya que presenta importantes ventajas:


Al igual que la PCR convencional, la qPCR consiste en la amplificación de un fragmento del genoma utilizando un par de oligonucleótidos, llamados cebadores, que son complementarios a dicha región. En la qPCR, este fragmento se detecta por fluorescencia tras cada ciclo (en “tiempo real”).

La gran diferencia a nivel genómico entre los genotipos I y II posibilita su detección específica en una única reacción de RT-qPCR utilizando diferentes parejas de cebadores y sondas marcadas con moléculas fluorescentes distintas. Del mismo modo puede incorporarse la detección adicional de controles internos.

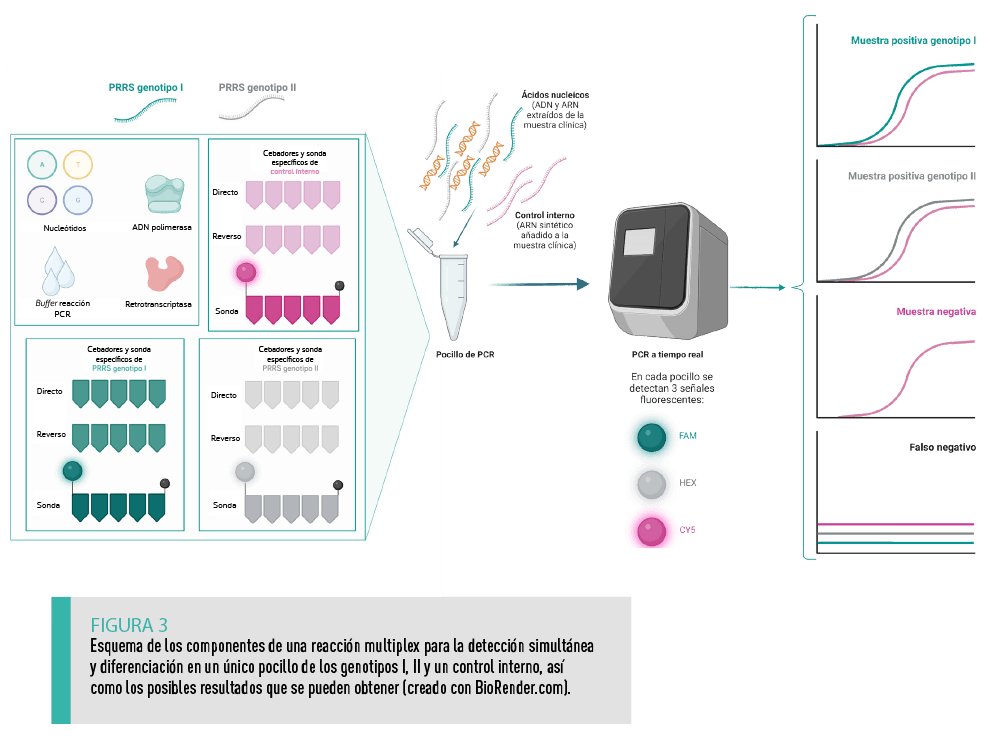
| Dentro de cada genotipo, el diseño de los cebadores y sondas está dirigido a regiones genómicas más conservadas entre las distintas cepas, como el ORF723. Sin embargo, el diagnóstico por RT-qPCR supone un reto constante dada la elevada tasa de mutación del virus PRRS. Se requiere un trabajo continuo de revisión y actualización de estos ensayos para asegurar la detección del mayor número de cepas circulantes. |
La capacidad de esta técnica para identificar regiones genómicas concretas ha permitido el diseño de ensayos específicos para detectar cepas de alta patogenicidad o virulencia.

Considerando que las estrategias de prevención se basan en gran parte en la vacunación con cepas vivas atenuadas25 y que los kits comerciales para la detección de PRRSV no diferencian entre cepas vacunales y cepas campo, es importante conocer si un resultado qPCR positivo se debe a la presencia de una cepa vacunal para poder tomar las medidas de manejo apropiadas.

SECUENCIACIÓN DE SANGER
![]()
La secuenciación de Sanger es una metodología que consiste en la determinación de los nucleótidos que conforman una región de ADN. Permite obtener secuencias de hasta 900-1000 pares de bases, lo que resulta muy útil para secuenciar genes o secuencias cortas.
| La secuenciación de Sanger del virus PRRS tiene aplicación directa en estudios epidemiológicos y en la mejora y actualización continua de los kits de qPCR de diagnóstico para no perder sensibilidad frente a nuevas cepas emergentes. |
LA SECUENCIACIÓN DE SANGER DEL VIRUS PRRS, EN CONCRETO DEL ORF5, ES UN ANÁLISIS DE RUTINA EN LA MAYORÍA DE LOS LABORATORIOS VETERINARIOS
ORF5
El ORF5, de una longitud de 606 bp, contiene regiones inmunogénicas y regiones hipervariables que posibilitan una discriminación entre las diferentes cepas27.

Una vez obtenida la secuencia final, es posible compararla con las secuencias ORF5 de las cepas vacunales o con otras cepas secuenciadas previamente en la misma explotación o pirámide productiva (Figura 4A y B).
Si bien, se trata de un valor arbitrario, está generalmente aceptado un cut-off de 97% de similitud en la secuencia de nucleótidos para el ORF5 para determinar que con alta probabilidad se trate de la misma cepa. No obstante, para llegar a esta conclusión, es importante considerar:
¿Qué nos dicen los resultados de la secuenciación de Sanger sobre la bioseguridad de la granja?
| Problema de bioseguridad interna Si se determina que se trata de la misma cepa que la detectada y secuenciada en un momento anterior, se sospechará de un problema de bioseguridad interna. |
Problema de bioseguridad externa Cuando no exista una elevada identidad nucleotídica con cepas anteriores, podrá atribuirse a un fallo en los sistemas de bioseguridad externa. |

De esta forma se podrán tomar las acciones correctivas más apropiadas
para disminuir las probabilidades de transmisión del virus.
ANÁLISIS FILOGENÉTICOS
Si la secuenciación se realiza de forma rutinaria y se dispone de secuencias a lo largo del tiempo y de una región geográfica, el análisis de las relaciones filogenéticas entre ellas da una imagen muy útil de la situación epidemiológica del virus en un área y su progresión y transmisión en un periodo de tiempo determinado (Figura 4C).

Es cierto que la secuenciación de ORF5 proporciona una valiosa información epidemiológica y tiene aplicación directa en campo. Sin embargo, no se puede pasar por alto que esta región representa un 5% del genoma de PRRS por lo que no permite captar toda la variabilidad genética de una cepa (Figura 1)7.
En este punto, cabe destacar los fenómenos de recombinación que se han descrito para ambos genotipos de PRRS y que pueden ocurrir a lo largo de diversos puntos en todo el genoma, incluso dentro de un mismo ORF.

Además, ante una infección por una cepa recombinante se puede realizar una asunción errónea de una recirculación si esta comparte una alta similitud en ORF5 con otras cepas detectadas previamente.
LAS DISCREPANCIAS ENTRE EL DIAGNÓSTICO Y LOS SIGNOS CLÍNICOS OBSERVADOS PUEDEN SER UNA PISTA PARA SOSPECHAR DE UNA CEPA RECOMBINANTE29
En estos casos, mediante secuenciación Sanger se pueden amplificar varios fragmentos del genoma que cubran una región más amplia (ORF2-5 u ORF2-7) o simplemente de otro ORF no adyacente al ORF5, por ejemplo, el ORF1a u ORF2, permitiendo identificar este tipo de cepas30.
Finalmente, es importante conocer las limitaciones de la secuenciación Sanger:


SECUENCIACIÓN MASIVA
![]()
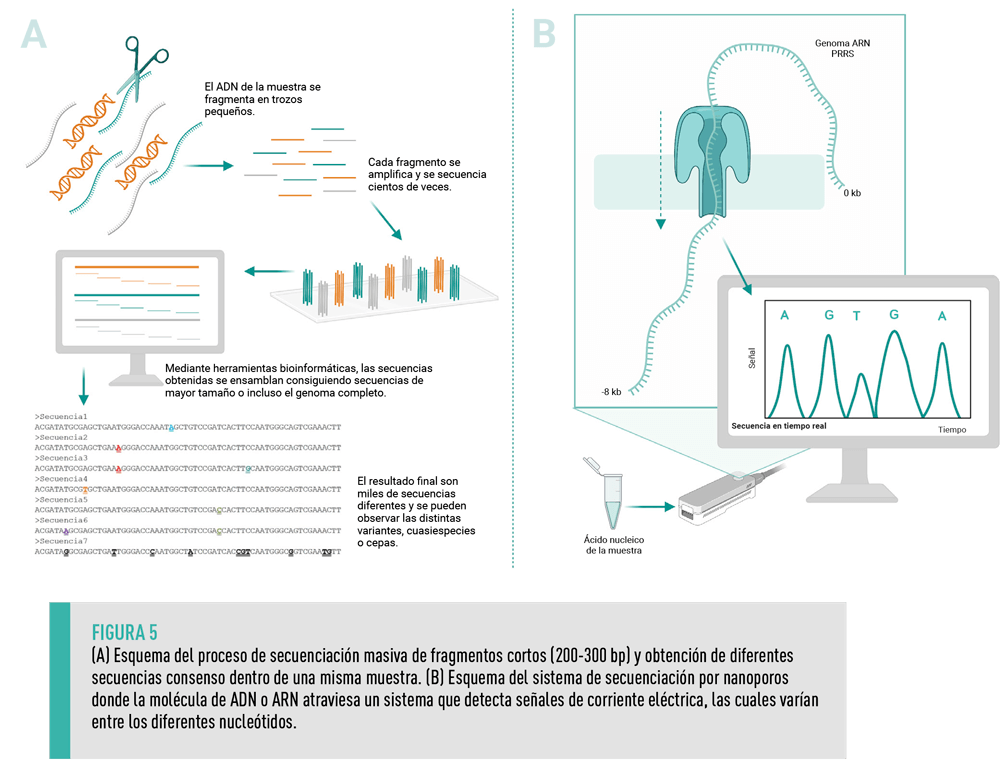
Comúnmente denominada NGS (del inglés Next Generation Sequencing), la secuenciación masiva permite la obtención miles de secuencias cortas, unos 200- 300 pares de bases, de forma paralela.

En comparación a la secuenciación de Sanger:



Recientemente, se han desarrollado nuevas tecnologías que ofrecen secuenciación de fragmentos mucho más largos (>10.000 pares de bases).
En su aplicación al estudio del PRRSV, la secuenciación masiva ha aumentado el número de genomas completos disponibles. Gracias a ello, se ha determinado que, aparte del ORF5, existen otras regiones con más variabilidad como el principio del ORF1a31.
CONOCER LA TOTALIDAD DEL GENOMA IMPLICA TENER UNA CEPA COMPLETAMENTE CARACTERIZADA Y ESTABLECER DE FORMA MUCHO MÁS PRECISA LAS RELACIONES FILOGENÉTICAS ENTRE CEPAS
| Además, impide una malinterpretación epidemiológica debido a los eventos de recombinación mencionados anteriormente y se pueden identificar marcadores epidemiológicos y/o detectar nuevas cepas emergentes24. |
Con la secuenciación masiva no se obtiene una única secuencia como en la secuenciación de Sanger, sino que todo el material genético en una muestra, donde pueden encontrarse distintas variantes o mutantes de PRRSV, se secuencia (Figura 5A).

A nivel de investigación, con la secuenciación masiva se puede ahondar, entre otros, en:
| Todo este conocimiento constituye la base para continuar mejorando el diagnóstico, las estrategias de control de la enfermedad y el diseño de nuevas vacunas que confieran mayor protección frente a las cepas circulantes32,33. |


OTRAS HERRAMIENTAS PARA EL DIAGNÓSTICO MOLECULAR DE PRRS
![]()
La rapidez en el diagnóstico para aplicar medidas correctivas en el menor tiempo posible es clave ante un brote de una enfermedad y el PRRS no es una excepción.
Existen técnicas en desarrollo para hacer diagnósticos in-situ sin la necesidad de equipos especializados. Entre otros sistemas, la PCR isoterma y la amplificación por recombinasa se han descrito como posibles herramientas para detectar el virus PRRSV con buena sensibilidad y especificidad.

No obstante, para poder explotar su potencial todavía se debe optimizar su sensibilidad y su robustez para que su resultado llegue a ser comparable a la qPCR o poder usar muestras clínicas directas sin extracción previa de ácidos nucleicos35,36.
Las últimas tecnologías de secuenciación desarrolladas (tercera generación) han conseguido ampliar la longitud de las secuencias obtenidas, así como poder secuenciar directamente moléculas ARN.
Una de estas tecnologías se basa en el uso de nanoporos (tecnología MiniON Oxford Nanopore) y se realiza con un pequeño dispositivo portable conectado a un ordenador (Figura 5B).

HAY QUE DESTACAR LA IMPORTANCIA DE REGISTRAR, ANALIZAR Y USAR TODA LA INFORMACIÓN QUE SE RECOPILA DE FORMA GLOBAL
Cada día, en los laboratorios veterinarios se realizan cientos de análisis moleculares de muchos patógenos, generando multitud de informes individuales. El análisis de la información diagnóstica desde una perspectiva global de una enfermedad como el PRRSV, incluso en combinación con otras enfermedades, proporciona información muy valiosa.


CONCLUSIONES
La RT-qPCR es una herramienta indispensable para el diagnóstico de PRRS de forma sensible y rápida ante un brote o como forma de monitorización. Sin embargo, debe ir de la mano de la secuenciación para proporcionar la información genética de la cepa detectada, indispensable para la toma de decisiones a nivel de granja.
ES IMPORTANTE QUE LA INTERPRETACIÓN DEL RESULTADO DE SECUENCIACIÓN SE REALICE JUNTO CON LA OBSERVACIÓN CLÍNICA Y LA SITUACIÓN EPIDEMIOLÓGICA DE LA EXPLOTACIÓN O LA REGIÓN
El seguimiento epidemiológico basado en la secuenciación de ORF5 resulta de gran utilidad, pero la secuenciación de otras regiones más amplias u ORFs no contiguos puede ampliar la información obtenida y confirmar presencia de cepas recombinantes.
La secuenciación masiva podrá en el futuro reemplazar a la secuenciación de ORFs individuales, brindando mucha más información y proporcionando una caracterización genética completa.
Asimismo, el desarrollo y la optimización de las nuevas tecnologías surgidas podrán ofrecer un diagnóstico más rápido y a pie de granja, algunas de ellas aportando información genética en tiempo real.
| Todo ello en conjunto y recopilando de forma adecuada la información en las bases de datos permitirá abrir nuevas vías para seguir avanzando en el control y conocimiento del PRRS. |
Te puede interesar: Diagnóstico de laboratorio de procesos infecciosos – Toma de muestras y técnicas
BIBLIOGRAFÍA
1. Neumann, E. J. et al. Assessment of the economic impact of porcine reproductive and respiratory syndrome on swine production in the United States. J Am Vet Med Assoc (2005) doi:10.2460/javma.2005.227.385.
2. Holtkamp, D. J. et al. Assessment of the economic impact of porcine reproductive and respiratory syndrome virus on United States pork producers. Journal of Swine Health and Production Preprint at (2013).
3. Nathues, H. et al. Cost of porcine reproductive and respiratory syndrome virus at individual farm level – An economic disease model. Prev Vet Med (2017) doi:10.1016/j.prevetmed.2017.04.006.
4. Albina, E. [Porcine reproductive and respiratory syndrome: ten years of experience (1986-1996) with this undesirable viral infection]. Vet Res (1997).
6. Forsberg, R. Divergence time of porcine reproductive and respiratory syndrome virus subtypes. Mol Biol Evol (2005) doi:10.1093/molbev/msi208.
7. Murtaugh, M. P., Stadejek, T., Abrahante, J. E., Lam, T. T. Y. & Leung, F. C. C. The ever expanding diversity of porcine reproductive and respiratory syndrome virus. Virus Research Preprint at https://doi.org/10.1016/j.virusres.2010.08.015 (2010).
8. Dokland, T. The structural biology of PRRSV. Virus Research Preprint at https://doi.org/10.1016/j. virusres.2010.07.029 (2010).
9. Domingo, E. & Schuster, P. What is a quasispecies? Historical origins and current scope. Curr Top Microbiol Immunol (2016) doi:10.1007/82_2015_453.
10. Lu, Z. H. et al. Quasispecies evolution of the prototypical genotype 1 porcine reproductive and respiratory syndrome virus early during in vivo infection is rapid and tissue specific. Arch Virol (2017) doi:10.1007/s00705-017-3342-0.
11.Shi, M. et al. Molecular epidemiology of PRRSV: A phylogenetic perspective. Virus Research Preprint at https://doi.org/10.1016/j.virusres.2010.08.014 (2010).
12. Pileri, E. & Mateu, E. Review on the transmission porcine reproductive and respiratory syndrome virus between pigs and farms and impact on vaccination. Veterinary Research Preprint at https://doi.org/10.1186/s13567-016-0391-4 (2016).
13. Drew, T. W. The emergence and evolution of swine viral diseases: To what extent have husbandry systems and global trade contributed to their distribution and diversity? OIE Revue Scientifique et Technique (2011) doi:10.20506/rst.30.1.2020.
14. Yaeger, M. J. The diagnostic sensitivity of immunohistochemistry for the detection of porcine reproductive and respiratory syndrome virus in the lung of vaccinated and unvaccinated swine. Journal of Veterinary Diagnostic Investigation (2002) doi:10.1177/104063870201400104.
15. Belák, S. Molecular diagnosis of viral diseases, present trends and future aspects. A view from the OIE Collaborating Centre for the Application of Polymerase Chain Reaction Methods for Diagnosis of Viral Diseases in Veterinary Medicine. Vaccine (2007) doi:10.1016/j.vaccine.2006.11.068.
16. Christopher-Hennings, J. et al. Detection of porcine reproductive and respiratory syndrome virus in boar semen by PCR. J Clin Microbiol (1995) doi:10.1128/ jcm.33.7.1730-1734.1995.
17. Kittawornrat, A. et al. Porcine reproductive and respiratory syndrome virus (PRRSV) in serum and oral fluid samples from individual boars: Will oral fluid replace serum for PRRSV surveillance? Virus Research Preprint at https://doi.org/10.1016/j.virusres.2010.07.025 (2010).
18. Rotolo, M. L. et al. Sampling guidelines for oral fluidbased surveys of group-housed animals. Vet Microbiol (2017) doi:10.1016/j.vetmic.2017.02.004.
19. Lopez, W. A., Angulo, J., Zimmerman, J. J. & Linhares, D. C. L. Porcine reproductive and respiratory syndrome monitoring in breeding herds using processing fluids. Journal of Swine Health and Production Preprint at (2018).
20. Baliellas, J., Novell, E., Enric-Tarancón, V., Vilalta, C. & Fraile, L. Porcine reproductive and respiratory syndrome surveillance in breeding herds and nurseries using tongue tips from dead animals. Vet Sci (2021) doi:10.3390/vetsci8110259.
21. Kleiboeker, S. B., Lehman, J. R. & Fangman, T. J. Concurrent use of reverse transcription-polymerase chain reaction testing of oropharyngeal scrapings and paired serological testing for detection of porcine reproductive and respiratory syndrome virus infection in sows. Journal of Swine Health and Production (2002).
22.Lurchachaiwong, W. et al. Rapid detection and strain identification of porcine reproductive and respiratory syndrome virus (PRRSV) by real-time RT-PCR. Lett Appl Microbiol (2008) doi:10.1111/j.1472-765X.2007.02259.x.
23.Wernike, K. et al. Detection and typing of highly pathogenic porcine reproductive and respiratory syndrome virus by multiplex real-time RT-PCR. PLoS One (2012) doi:10.1371/journal.pone.0038251.
24. Liu, Y. et al. Emergence of Different Recombinant Porcine Reproductive and Respiratory Syndrome Viruses, China. Sci Rep (2018) doi:10.1038/s41598-018-22494-4.
25. Linhares, D. C. L. et al. Effect of modified-live porcine reproductive and respiratory syndrome virus (PRRSv) vaccine on the shedding of wild-type virus from an infected population of growing pigs. Vaccine (2012) doi:10.1016/j.vaccine.2011.10.075.
26. Rawal, G. et al. Development and validation of a reverse transcription real-time PCR assay for specific detection of PRRSGard vaccine-like virus. Transbound Emerg Dis 69, 1212–1226 (2022).
27. Díaz, I. et al. In silico prediction and ex vivo evaluation of potential T-cell epitopes in glycoproteins 4 and 5 and nucleocapsid protein of genotype-I (European) of porcine reproductive and respiratory syndrome virus. Vaccine (2009) doi:10.1016/j.vaccine.2009.07.029.
28. Nan, Y. et al. Improved vaccine against PRRSV: Current Progress and future perspective. Frontiers in Microbiology Preprint at https://doi.org/10.3389/fmicb.2017.01635 (2017).
29. Risser, J. et al. Porcine reproductive and respiratory syndrome virus genetic variability a management and diagnostic dilemma. Virology Journal Preprint at https://doi.org/10.1186/s12985-021-01675-0 (2021).
30. Vandenbussche, F., Mathijs, E., Tignon, M., Vandersmissen, T. & Cay, A. B. Wgs versus orf5 based typing of prrsv: A belgian case study. Viruses (2021) doi:10.3390/v13122419.
31. Lu, Z. H., Archibald, A. L. & Ait-Ali, T. Beyond the whole genome consensus: Unravelling of PRRSV phylogenomics using next generation sequencing technologies. Virus Res (2014) doi:10.1016/j.virusres.2014.10.004.
32. Cortey, M., Díaz, I., Martín-Valls, G. E. & Mateu, E. Next-generation sequencing as a tool for the study of Porcine reproductive and respiratory syndrome virus (PRRSV) macro- and micro- molecular epidemiology. Veterinary Microbiology Preprint at https://doi.org/10.1016/j.vetmic.2017.02.002 (2017).
33. Dortmans, J. C. F. M., Buter, G. J., Dijkman, R., Houben, M. & Duinhof, T. F. Molecular characterization of type 1 porcine reproductive and respiratory syndrome viruses (PRRSV) isolated in the Netherlands from 2014 to 2016. PLoS One (2018) doi:10.1371/journal.pone.0218481.
34. Gagnon, C. A., Lalonde, C. & Provost, C. Porcine reproductive and respiratory syndrome virus wholegenome sequencing efficacy with field clinical samples using a poly(A)-tail viral genome purification method. Journal of Veterinary Diagnostic Investigation (2021) doi:10.1177/1040638720952411.
35. Park, J. Y. et al. Reverse-transcription loop-mediated isothermal amplification (RT LAMP) assay for the visual detection of European and North American porcine reproductive and respiratory syndrome viruses. J Virol Methods (2016) doi:10.1016/j.jviromet.2016.08.008.
36. Tian, X. X. et al. Rapid visual detection of porcine reproductive and respiratory syndrome virus via recombinase polymerase amplification combined with a lateral flow dipstick. Arch Virol (2022) doi:10.1007/s00705-021-05349-8.
37. Tan, S., Dvorak, C. M. T. & Murtaugh, M. P. Rapid, unbiased PRRSV strain detection using MinION direct RNA sequencing and bioinformatics tools. Viruses (2019) doi:10.3390/v11121132.
38. Anderson, T. K. et al. The United States Swine Pathogen Database: integrating veterinary diagnostic laboratory sequence data to monitor emerging pathogens of swine. Database (2021) doi:10.1093/database/baab078.
[/registrados]
